Forschungsbericht 2021 - Max-Planck-Institut für Pflanzenzüchtungsforschung
Modellierung von Pflanzenentwicklung und Artenvielfalt
Explodierende Samenkapseln
Die hier vorgestellten Studien stützen sich auf zwei verwandte Arten, Cardamine hirsuta und Arabidopsis thaliana, um im Vergleich zu verstehen, wie sich die Formen der beiden verschiedenen Arten entwickeln und diversifizieren [1]. Trotz ihres hohen Verwandtheitsgrads ist die Ausprägung ihrer jeweiligen Morphologie unterschiedlich.
Explosionen von Samenkapseln gehören zu den schnellsten Bewegungen im Pflanzenreich. Die Samen des kleinen Wildkrauts C. hirsuta werden mit einer erstaunlichen Geschwindigkeit von über 10 Metern pro Sekunde ausgestoßen. In weniger als 3 Millisekunden schleudern die sich schnell aufrollenden Fruchtklappen ihre Samen aus. Diese Ausbreitungsstrategie unterscheidet sich deutlich von der nichtexplosiven Öffnung der A. thaliana Samenkapseln und ist ein wirksames Mittel, um die Samen effektiv zu verteilen. Um dies zu erreichen, erfolgt die Speicherung und schnelle Freisetzung von elastischer Energie auf der Ebene von Zellen und Geweben. Wir haben experimentelle Daten auf diesen Ebenen durch Computermodelle verknüpft [2]. Damit haben wir ein Multiskalenmodell geschaffen, das die explosive Samenausbreitung reproduzieren kann. Zur Validierung unseres Ansatzes konnten wir zeigen, dass die verschiedenen Modelle auf allen Ebenen konsistente Vorhersagen liefern.
Nun gilt es, diejenigen Gene in C. hirsuta zu identifizieren, die erforderlich sind, um explodierende Samenkapseln auch in einer Pflanze wie A. thaliana zu rekonstruieren.
Rekonstruktion von Blattformen
Eine Vorhersage von eins-zu-eins-Beziehungen zwischen Genotypen und Phänotypen zu treffen, kann schwierig sein. Dies liegt daran, dass viele Gene in komplexer Wechselwirkung miteinander einen einzelnen Phänotyp beeinflussen können. Noch schwieriger ist es, vorherzusagen, wie Genotypen im Laufe der Entwicklung in Phänotypen umgesetzt werden. Viele verschiedene Wachstumsmuster können die gleiche Form hervorbringen. Daher ist es von entscheidender Bedeutung, den Umfang, die Dauer und die Richtung des Zellwachstums zu messen, um zu verstehen, wie Gene die Form ausprägen.
Dafür haben wir eine neue Live-Bildgebungsmethode entwickelt, die es uns ermöglicht, das Wachstum mit zellulärer Auflösung über die gesamte Blattoberfläche zu quantifizieren; vom Blattaustrieb bis zu seiner charakteristischen Form 5 bis 8 Tage später (Abb. 1). Keine leichte Aufgabe, da die kritische Wachstumszone und die strukturgebende Region entlang des Blattrandes tief unter den benachbarten Blättern an der wachsenden Triebspitze verborgen sind. Wir nutzten diese Methode, um einen umfassenden Datensatz mit genauen Messungen des Wachstums und der Genexpression auf Zellebene als Reaktion auf verschiedene genetische und pharmakologische Störungen bei A. thaliana und C. hirsuta zu erstellen.
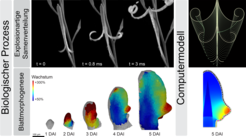
Zur Analyse des Datensatzes erstellten wir Karten einzelner „Blattschicksale“ und verglichen den Grad der Erhaltung beziehungsweise Divergenz der Eigenschaften an entsprechenden Stellen in wachsenden Blattanlagen. Dieser neue Ansatz ist im Wesentlichen das entwicklungsbiologische Äquivalent eines DNA-Sequenzvergleichs: Statt Nukleotide an äquivalenten Positionen in verschiedenen Gen-Sequenzen zu vergleichen, werden Zellwachstumsparameter an äquivalenten Positionen in verschiedenen Blattanlagen verglichen. Wir bezeichnen dieses Analyseinstrument als Wachstums-Alignment-Graph.
Beim Übergang vom Experiment zur Theorie konstruierten wir das erste physikalisch basierte Rechenmodell zur Blattmorphogenese von A. thaliana (Abb. 1). In diesem Modell ergibt sich die Blattform aus einem genetisch kontrollierten zellulären Verhalten. Ein interdisziplinärer Ansatz wie dieser vereint die Vorteile aus beiden Welten: Einerseits liefert die Modellierung mechanistische Erkenntnisse, und andererseits liefern Daten aus der direkten Live-Bildgebungsbeobachtung genügend Informationen, um eine biologische Bedeutung zu finden und die Modelle mit realen Genaktivitäten zu verknüpfen.
Die Blattformen von A. thaliana und C. hirsuta unterscheiden sich auffallend - die eine ist einfach, die andere zergliedert. Erstaunlicherweise haben wir festgestellt, dass das Wachstum der frühen Blattanlagen bei beiden Arten aber ähnlich verläuft. Ihre unterschiedlichen Blattformen entstehen aus dem Zusammenspiel zwischen einem Standardwachstumsmodus und einem Mustergenerator am Blattrand. Wir konnten zwei Homeobox-Gene identifizierten, REDUCED COMPLEXITY (RCO) und SHOOT MERISTEMLESS (STM), die das Wachstum lokal (RCO) und global (STM) im Blatt regulieren. Durch die Beeinflussung des Zusammenspiels zwischen dem Standardwachstumsmodus und der Randmusterung gestalten diese beiden Gene die Blattvielfalt.
Prädiktive Biologie
In einem transgenen Experiment rekonstruierten wir die Blattform. Wir verwendeten RCO und STM als die minimalen Komponenten für die sezierte Blattform. Als wir diese Gene in geeignete Bereiche des Arabidopsis-Blattes einführten, konnten wir wichtige Aspekte der Blattform von C. hirsuta rekonstruieren (Abb. 2). Unser Ansatz ermöglichte es uns also, ein Gewebemerkmal in seine Bestandteile auf Zellebene zu zerlegen und es dann zu übertragen. Die Vorteile eines solchen Ansatzes sind groß. Durch die Definition weniger Komponenten und das Verständnis, warum bestimmte Merkmale der Blattgeometrie rekonstruiert werden konnten, haben wir ein beträchtliches Maß an Vorhersagbarkeit in unserem Verständnis der Blattentwicklung und -vielfalt erreicht.

Diese Schritte hin zu einer vorhersagbaren Biologie tragen dazu bei, von Neugierde getriebene Wissenschaft voranzubringen. Die prädiktive Biologie ist auch ein gemeinsames Ziel, die Wissenschaft zum Nutzen der Gesellschaft voranzubringen, zum Beispiel durch die Verbesserung der Effizienz und Nachhaltigkeit in der Pflanzenproduktion.













