Schnelle Mikroskope mit hoher Auflösung
Optimierte DNA-Sequenzen ermöglichen schnellere Bildaufnahme mit DNA-PAINT
DNA-PAINT nutzt DNA-basierte Sonden, um kleinste zelluläre Strukturen sichtbar zu machen. Bislang war die Technik durch vergleichsweise langsame Bildaufnahme limitiert. Optimiertes DNA-Sequenzdesign erlaubt es, nun Ergebnisse zehnfach schneller zu erhalten und ebnet den Weg zu Hochdurchsatzstudien mit biomedizinischer Relevanz.

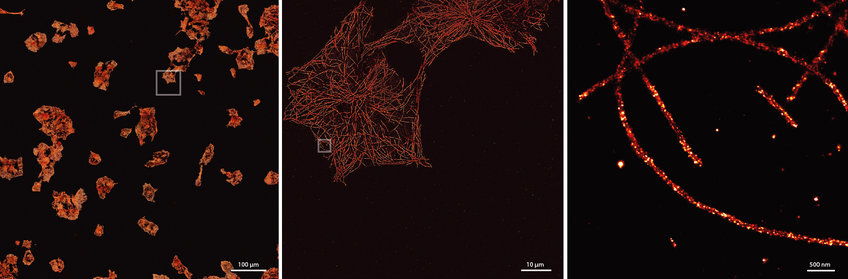
Fortschritte in der Fluoreszenzmikroskopie ermöglichen es, biologische Prozesse unterhalb der klassischen Beugungsgrenze des Lichtes sichtbar zu machen. Eine Variante dieser sogenannten Superauflösungstechniken ist DNA-PAINT, die Ralf Jungmann, Forschungsgruppenleiter am Max-Planck-Institut für Biochemie und Professor für Experimentalphysik an der Ludwig-Maximilians-Universität München, mit seinen Kollegen entwickelt hat. „Mit der DNA-PAINT können wir superaufgelöste Bilder mit technisch vergleichsweise einfachen Mikroskopen erhalten“, sagt Jungmann. Um das zur Rekonstruktion superaufgelöster Bilder notwendige „Blinken“ von Zielmolekülen zu erreichen, werden diese bei DNA-PAINT mit kurzen DNA-Strängen markiert. In Lösung befindet sich ein komplementärer, farbstoffmakrierter DNA-Strang, der wiederholt an den Zielstrang an- und abbindet und das Ziel zum „Blinken“ bringt. So erreicht DNA-PAINT sehr hohe Ortsauflösungen von weniger als zehn Nanometern und kann durch die Nutzung verschiedener DNA-Sequenzen, quasi Barcodes, viele Zielmoleküle gleichzeitig abbilden.
„In den letzten Jahren haben wir die Technik in vielen Bereichen verbessert. Eine große Einschränkung hat uns jedoch immer daran gehindert DNA-PAINT für biologisch relevante Hochdurchsatzstudien einzusetzen: Die vergleichsweise langsame Bildaufnahme“, sagt Jungmann. Klassische DNA-PAINT-Experimente nehmen üblicherweise mehrere zehn Minuten bis hin zu Stunden in Anspruch. „Wir haben uns genau angeschaut, warum das so lange dauert“, so Florian Schüder, Erstautor der Studie und Mitarbeiter im Labor von Jungmann. „Durch optimiertes DNA-Sequenzdesign und verbesserte Pufferbedingungen konnten wir die Geschwindigkeit um einen Faktor Zehn erhöhen“, so Schüder weiter.
Einfluss der DNA-Sequenz auf Hybridisierung

Hierzu haben die Forscher in Kollaboration mit der Abteilung von Petra Schwille den Einfluss der DNA-Sequenz und der Basenabfolge auf die Geschwindigkeit der Ausbildung der Doppelhelix untersucht. Der farbstoffmarkierte Einzelstrang, bei DNA-PAINT auch ‚Imager‘ genannt, besteht generell aus den Grundbausteinen der DNA, den vier Basen: Adenin (A), Thymin (T), Guanin (G) und Cytosin (C). Der Einfluss der Basenkomposition und Länge der Einzelstränge auf die Bindezeit ist recht gut verstanden: Je länger der Doppelstrang und je mehr GC-Basenpaare in der Sequenz vorhanden sind, desto stabiler ist der DNA-Duplex und desto länger die Bindezeit. Der Einfluss auf die Geschwindigkeit der Duplexbildung, und damit die Blinkgeschwindikgeit in DNA-PAINT, ist jedoch deutlich schlechter untersucht.
Hier konnten die Forscher nun zeigen, dass die Ausbildung intramolekularer Haarnadelkonformationen, also das Falten mit sich selbst, in kurzen Strängen durch die Reduktion auf zwei Basen (z.B. nur T und C statt A, T, C, G) vollständig unterbunden werden kann. „Wir haben jetzt die Stränge so entworfen, dass wir nur ein sogenanntes ‚Zweibuchstabenalphabet‘, z.B. nur T und C oder nur A und G, benutzen. So konnten wir die Bindefrequenz bereits um einen Faktor Fünf steigern“, erklärt Schüder. „Durch weitere Optimierungen am verwendeten Puffersystem konnten wir zusätzlich einen Faktor Zwei rausholen, so dass wir jetzt zehnmal schneller Bilder aufnehmen können."
Von der DNA-Origami-Testplattform zur Zelle
Um die Verbesserungen der DNA-PAINT-Technik zu testen, kombinierten die Forscher diese mit DNA-Origami-Strukturen. Hierbei handelt es sich um selbstassemblierende DNA-Objekte in Nanometergröße, die sich autonom zu einer Art „Steckplatte“ falten. Auf dieser befinden sich die Gegenstränge, die als definierte Punkte im Abstand von ungefähr fünf Nanometern aufgetragen sind. Unter dem Mikroskop untersuchten die Forscher so die verbesserte Messgeschwindigkeit unter definierten Bedingungen. Im nächsten Schritt konnte die verbesserte Aufnahmegeschwindigkeit auch in zellulärer Umgebung gezeigt werden. Hierzu wurden in einer Zellprobe die Mikrotubli, ein Teil des Zytoskelettes, mit zehnfach höherer Geschwindigkeit sichtbar gemacht. „Wir konnten so ein quadratmillimetergroßes Areal in acht Stunden bei einer Auflösung von 20 Nanometern aufnehmen. Das hätte vorher fast vier Tage gedauert“, erklärt Florian Schüder.
Ralf Jungmann fasst zusammen: „Mit der in dieser Studie gezeigten höheren Abbildungsgeschwindigkeit eröffnen wir ein neues Kapitel superaufgelöster DNA-basierter Mikroskopie. Dies sollte es uns nun erlauben, DNA-PAINT für Hochdurchsatzstudien mit biologischer und biomedizinischer Relevanz z.B. in der Diagnostik einzusetzen.“