Forschungsbericht 2007 - Max-Planck-Institut für terrestrische Mikrobiologie
Regulation des Verlaufs der Entwicklung bei Myxococcus xanthus
Regulation of developmental progression in Myxococcus xanthus
Ökophysiologie (Søgaard-Andersen) (Prof., M.D., Ph.D. Lotte Søgaard-Andersen)
MPI für terrestrische Mikrobiologie, Marburg
Einleitung
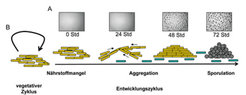
Myxococcus xanthus ist ein ubiquitär verbreitetes, stäbchenförmiges Bodenbakterium, das durch einen komplexen Lebenszyklus und einer Art sozialem Verhalten gekennzeichnet ist. In nährstoffreicher Umgebung findet vegetatives Wachstum statt. Werden hingegen Nährstoffe knapp, wird ein Entwicklungszyklus initiiert [1].
Bei vegetativem Wachstum erwerben M. xanthus -Zellen Nährstoffe kooperativ, indem sie über Oberflächen schwärmen und dabei Antibiotika und Abbauenzyme freisetzen, um Beuteorganismen zu lähmen und zu verdauen oder um organisches Material abzubauen. Die Wachstumsrate der Zellen hängt von der Populationsdichte ab und es ist für die Zellen von Vorteil, Gruppen zu bilden. Für einen alternativen Entwicklungszyklus ist dagegen die Bildung von Fruchtkörpern charakteristisch. Diese Fruchtkörper sind multizelluläre kuppelförmige Strukturen von ungefähr 40 µm Höhe. Sie sind mit metabolisch inaktiven Sporen gefüllt, die auch längere Zeiträume ohne Nährstoffzufuhr überdauern können. Die Zellen beginnen das Entwicklungsprogramm, sobald zellintern ein Mangel an Aminosäuren festgestellt wird. Daraufhin teilt sich die Zellpopulation in mindestens drei Subpopulationen mit jeweils unterschiedlicher Bestimmung. Eine dieser Subpopulationen, die etwa 80 % aller Zellen zu Beginn des Zyklus umfasst, lysiert. Dies geschieht vermutlich, um den übrigen Zellen genug Nährstoffe zur Vollendung des Entwicklungsyklus zur Verfügung zu stellen. Von den überlebenden Zellen sind 70 % an der Bildung des Fruchtkörpers beteiligt, die übrigen 30% verbleiben außerhalb der Fruchtkörper als so genannte periphere Stäbchen. Diese differenzieren nicht in Sporen, unterscheiden sich jedoch von vegetativ wachsenden Stäbchen. Es wird angenommen, dass sie während des Entwicklungsprogramms eine Rolle bei der Wahrnehmung von Änderungen in der Umgebung spielen.
Der überwiegende Teil der bisherigen Forschung konzentrierte sich auf das komplexe Verhalten der Zellen, die an der Fruchtköperbildung beteiligt sind. Zu Beginn des Entwicklungsprogramms aggregieren bewegliche Zellgruppen zu hügelartigen Strukturen von jeweils ca. 105-106 Zellen (Abb. 1). Erst nach deren Ausbildung differenzieren sich Zellen innerhalb dieser Strukturen zu Sporen. Die Fruchtkörperbildung hat den Vorteil, dass auf diese Weise ganze Sporengruppen in neue, nährstoffreichere Umgebungen gelangen können, sodass nach dem Auskeimen sofort eine größere Zellpopulation entsteht, die effektiv neue Nährstoffe erschließen kann. Das Entwicklungsprogramm wird durch Genexpressionskaskaden und Proteinmodifikationen gesteuert. Diese werden durch eine zeitlich festgelegte Folge von intra- und interzellulären Signalen induziert, welche wiederum integriert und abgestimmt werden müssen, um das Verhalten sowohl einzelner Zellen als auch von Zellgruppen zu koordinieren [1].
Signaltransduktion in Myxococcus xanthus
M. xanthus enthält das umfangreichste Repertoire an Signalproteinen, das bisher in Bakterien beschrieben wurde. Darunter befindet sich eine besonders große Anzahl an Proteinen, die zu den Familien der Zwei-Komponenten-Systeme (two-component signaling, TCS) und der Protein-Serin/Threonin-Kinasen (PSTK) mit mindestens 272 beziehungsweise 102 Vertretern gehören [2, 3].
TCS-Systeme sind in Bakterien der vorherrschende Mechanismus zur Messung von und Reaktion auf Änderungen der Umgebung [4]. Das Modellsystem besteht aus zwei Proteinen. Dabei ist das erste Protein eine Sensor-Histidin-Protein-Kinase (HPK) mit einer Sensordomäne und einer konservierten Transmitterdomäne. Das zweite Protein wird als Response-Regulator (RR) bezeichnet und enthält eine konservierte Phosphoakzeptordomäne (receiver domain) und eine Reaktionsdomäne (output domain). Sobald die HPK ein Signal erhält, wird ein konservierter Histidinrest in der Transmitterdomäne autophosphoryliert. Diese Phosphogruppe wird anschließend an ein konserviertes Aspartat in der Phosphoakzeptordomäne des RR übertragen. Dadurch erfolgen die Aktivierung der Reaktionsdomäne und die Induktion einer entsprechenden Antwort, die typischerweise die Regulation bestimmter Genexpressionen ist. Obwohl die Mehrzahl der TCS-Systeme dem einfachen Zwei-Komponenten-Mechanismus unterliegt, können nach demselben Paradigma auch komplexere Systeme aufgebaut werden, da Transmitter- und Akzeptor-Domänen modular sind. In einem typischen einfachen System werden eine einzelne HPK und der dazugehörige RR zusammen in einer Transkriptionseinheit kodiert. Allerdings können in einigen Fällen HPK und RR separat vorliegen (orphans), wodurch die Identifikation des entsprechenden Partners in der Signalkaskade erschwert wird.
Im Gegensatz zu TCS-Systemen erfolgt in PSTK-Signaltransduktionssystemen die Weitergabe des Signals durch Phosphorylierung eines Serin- und/oder Threoninrestes (Ser/Thr) in einer konservierten Kinasedomäne. Die Aktivierung der Kinase erfolgt dabei entweder durch Autophosphorylierung oder durch die Aktivität einer weiteren, interagierenden Kinase. Die aktivierte Kinase phosphoryliert anschließend ein Zielprotein ebenfalls an einem Ser/Thr-Rest, was zur Modifikation der Aktivität des Zielproteins führt. PSTKs können allein oder zusammen mit anderen Kinasen in Phosphorylierungskaskaden oder Proteinkomplexen agieren, die bei der Regulierung des Signalflusses behilflich sind.
Aufgrund der Labilität der Phospho-Histidin/Aspartat-Gruppen wird vermutet, dass TCS-Proteine primär an der Weiterleitung kurzlebiger Signale beteiligt sind. Demgegenüber könnten PSTK-Signalsysteme zu langlebigeren Signalen und entsprechender Signalverstärkung führen, beruhend auf der deutlich höheren Stabilität von Phospho-Serin/Threonin-Gruppen. Interessanterweise wirken in M. xanthus einige TCS- und PSTK-Systeme bei der Regulation des Entwicklungsprogramms zusammen [5, 6].
Signalsysteme, die den Ablauf der Entwicklung regulieren
Mehrere Mutanten von M. xanthus wurden beschrieben, bei denen der Entwicklungszyklus schneller als beim Wildtyp abläuft. Dies resultiert in der Bildung kleinerer, zahlreicher und ungeordneter Fruchtkörper. Auffallenderweise befinden sich die Mutationen ausnahmslos in Genen, die für HPK-Proteine kodieren. Diese werden als espA und espC (early sporulation), red (regulation of early development) und todK (timing of development) bezeichnet [7, 8, 9, 10]. Die Kinasen weisen besondere Eigenschaften auf. Zunächst sind die genannten Proteine alle als orphans kodiert. Zweitens zeigen alle Kinasen eine untypische Anordnung von Sensor- oder Signalweiterleitungsdomänen. Dies lässt vermuten, dass sie an einem neuartigen Signaltransduktionsmechanismus beteiligt sind. Vermutlich ist der Ablauf des Entwicklungszyklus einer Steuerung durch einen oder mehrere negative Regulatoren unterworfen. Diese fungieren als Kontrollpunkt und sind notwendig für den koordinierten Ablauf und das zelluläre „Sozialverhalten“. Zudem repräsentieren diese Kontrollpunkte neuartige Mechanismen, die mehrere Signale zeitlich integrieren und koordinieren und damit das Sozialverhalten während der Entwicklung erst ermöglichen.
Aber arbeiten diese Kinasen zusammen, um den Ablauf des Entwicklungsprogramms zu regulieren? Sorgfältige vergleichende Analysen des Entwicklungszyklus jeder der Kinase-Mutanten lassen vermuten, dass Red früher im Entwicklungsprogramm agiert als TodK, welches wiederum früher wirkt als EspA/EspC, die ihrerseits phänotypisch identisch sind. Genetische Epistasis-Analysen, in denen die Entwicklung aller Kombinationen der Mutanten mit der der Einzelmutanten verglichen wurde, zeigten, dass mit Ausnahme von espA und espC alle anderen Kombinationen von Doppel-, Dreifach- und Vierfachmutanten in sich addierenden Phänotypen resultierten, wobei die Entwicklung zunehmend schneller ablief. Diese Analysen deuten darauf hin, dass drei genetisch distinkte Signalwege, bestehend aus EspA/EspC, Red und TodK, existieren, die den Ablauf der Entwicklung kontrollieren,.
Diese genetischen Analysen zeigten ebenfalls eine Korrelation zwischen der Geschwindigkeit des Entwicklungsablaufs und des Verlustes der Fähigkeit, geordnete Fruchtkörper zu bilden. Die sich am schnellsten entwickelnden Mutanten brachten nur flache, missgestaltete Fruchtkörper hervor und viele Sporen bildeten sich außerhalb dieser Strukturen (Abb. 2): Zellen, bei denen espA, espC, red und todK deletiert waren, aggregierten und sporulierten mehr als 30 Stunden früher als der Wildtyp und bildeten nur flache Sporenschichten anstelle abgegrenzter Fruchtkörper. Eine Untersuchung der Größenverhältnisse der Subpopulationen wies aber darauf hin, dass die Anzahl der Sporen in den Fruchtkörpern, so missgestaltet sie auch waren, der des Wildtyps entsprach. Allerdings nahm die Anzahl der peripheren Stäbchen in sich schneller entwickelnden Mutanten ab, während die Anzahl der sich außerhalb der Fruchtkörper beobachteten Sporen zunahm. Dies deutet darauf hin, dass auch die peripheren Stäbchen sporulierten. In diesem Falle wären die Kontrollpunkt-Systeme auch für die korrekte Festlegung des Zellschicksals entscheidend.
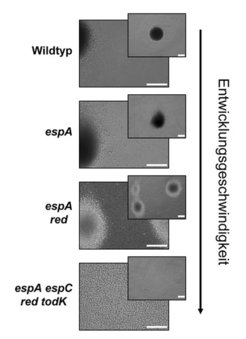
Wie agieren diese Kinasen als Kontrollpunkte der Entwicklung in Anbetracht der ungewöhnlichen Domänenanordnung innerhalb der Proteine? Wie wird das Signal weitergegeben? Der Fortschritt in der Charakterisierung zweier dieser Signalsysteme, EspA und Red, soll im Folgenden beschrieben werden.
EspA
EspA ist eine einzeln kodierte (orphan) Hybrid-HPK, welche einen ungewöhnlichen Sensor aufweist, bestehend aus einer FHA- (fork head associated) Domäne und zwei aufeinander folgenden PAS-Domänen. FHA-Domänen treten häufig in PSTK-Signalsystemen auf und vermitteln Proteininteraktionen durch Bindung an spezifische Phospho-Threonin enthaltende Proteine. Interessanterweise ist EspA eine der wenigen HPKs, die eine FHA-Domäne enthalten. Demgegenüber sind PAS-Domänen sehr häufig mit HPKs assoziiert und generell an der Messung des Energiestatus der Zelle beteiligt.
Das espA Gen ist in einer Genregion mit drei weiteren Genen espB, pktA5 und pktB8 lokalisiert, die ein mutmaßliches Membrantransportprotein und zwei PSTK-Proteine kodieren. Im Gegensatz zum sich vorzeitig entwickelnden Phänotyp einer espA-Mutante weisen Einzelmutanten in espB, pktA5 und pktB8 ebenso wie eine Dreifachmutante eine verzögerte Entwicklung auf [6, 7]. Eine Kombination genetischer Epistasis-Experimente und Untersuchungen zu Protein-Protein-Interaktionen lasssen vermuten, dass EspB/PktA5/PktB8 zusammen ein Signalmodul darstellen, das mit EspA über die FHA-Domäne interagiert. Es ist jedoch bisher unbekannt, welche Signale diese Proteine wahrnehmen. Zusammengenommen deuten die Ergebnisse auf folgendes Modell für EspA-vermittelte Kontrolle während des Verlaufs der Entwicklung: Früh während des Entwicklungsprogramms, nachdem die Zellen das Hungersignal wahrgenommen haben, jedoch noch vor der Induktion der Aggregation, autophosphoryliert EspA und transferiert die Phoshogruppe zur entsprechenden Empfängerdomäne, was zur Hemmung des weiteren Verlaufs der Entwicklung führt. In Reaktion auf ein oder mehrere bisher unbekannte Signale wird dann durch EspB/PktA/PktB8 EspA veranlasst, die Inhibierung der Entwicklung aufzuheben.
Während nach wie vor unbekannt ist, wie genau EspA den Verlauf der Entwicklung inhibiert, lassen Analysen bestimmter stadienspezifischer Markergene vermuten, dass EspA die Anreicherung von MrpC reprimiert. Bei MrpC handelt es sich um einen Schlüsselregulator, der zur Induktion der Zellaggregation nötig ist. Damit partizipiert EspA an einem äußerst ungewöhnlichen Signaltransduktionssystem, das sowohl TCS- als auch PSTK-Signalmechanismen beinhaltet und das durch Integration mehrerer Signale für einen koordinierten Ablauf der Entwicklung notwendig ist. Die zukünftige Aufgabe besteht in der Identifizierung des entsprechenden Response-Regulators von EspA, sofern ein solcher existiert. Ferner sollen die durch EspA und das EspB/PktA5/PktB8-Modul aufgenommenen Signale identifiziert werden. Schließlich soll eine Analyse der spezifischen Protein-Protein-Interaktionen zwischen diesen vier Proteinen erfolgen, welche letztendlich EspA zur Aufhebung der Hemmung der MrpC-Akkumulation veranlasst.
RedCDEF
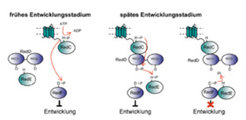
Das red (regulation of early development) Signal-Transduktionssystem ist ein weiteres Beispiel für einen komplexen Signalmechanismus. Es besteht aus zwei HPK-Proteinen (RedC und RedE) und zwei RR-Proteinen (RedD und RedF), die jedoch keine Reaktionsdomänen aufweisen. Die Gene, die für die genannten Proteine kodieren, werden gemeinsam als red-Operon transkribiert [8]. Die Ergebnisse einer Kombination von in vitro-Phosphorylierungs- und in vivo-Mutantenanalysen sowie Epistasis-Experimenten lassen den Schluss zu, dass die vier Red-Proteine zusammen an einem neuartigen Signalweiterleitungssystem beteiligt sind. Ein entsprechendes Modell ist in Abbildung 3 dargestellt. Während der frühen Entwicklungsphase, nach dem Empfang des Hungersignals und vor der Aggregation wird der RedF RR durch die HPK RedC phosphoryliert und der Fortgang des Entwicklungszyklus wird inhibiert. Der RR RedD und die HPK RedE bleiben dabei zunächst von den anderen Komponenten getrennt. In einer späteren Phase der Entwicklung nimmt der RR RedD ein bisher unbekanntes Signal auf und wird damit für die HPK RedC zugänglich, wodurch der RR RedF verdrängt wird. Daraufhin phosphoryliert die HPK RedC, den RR RedD, und die Phosphorylgruppe wird auf die HPK RedE übertragen. Folglich wird die HPK RedC aktiviert, um auf die ungewöhnlich stabile phosphorylierte Form von RedF als Phosphatase zu wirken, woraufhin die Inhibierung der Entwicklung aufgehoben wird. Die neuartigen Eigenschaften dieses Systems sind i.), dass RedC-F ein „Vier-Komponenten-Singnaltransduktionssystem“ bilden, dass ii.) der RR RedD notwendig ist, um die HPK RedC als Kinase für RedD zu aktivieren, iii.), dass die HPK RedE offensichtlich nicht autophosphoryliert, sondern stattdessen eine Phosphogruppe von RedD empfängt, und dass iv.) die HPK RedE ausschließlich als Phosphatase für RedF wirkt. Wie beim EspA-Signalsystem bestehen auch hier die zukünftigen Aufgaben in der Identifikation des Signals, des Mechanismus’ der Signalübertragung sowie der Zielproteine der Signalweiterleitung. Hierbei ermöglicht die Charakterisierung der Signalweiterleitung zwischen den Proteinen eine gezielte Suche nach den noch fehlenden Komponenten.
Abschließende Bemerkungen
Aufgrund des bemerkenswerten multizellulären Entwicklungsprogramms von M. xanthus und der großen Anzahl an Signal-Transduktionsproteinen stellt dieser Organismus ein hervorragendes System dar, um die Mechanismen der Signalweiterleitung und -integration zu untersuchen, die zur Koordination komplexen multizellulären Verhaltens nötig sind. Die Charakterisierung von Mutanten, die die Koordination der Fruchtkörperbildung verloren haben, zeigt, dass der Ablauf des Entwicklungsprogramms durch komplexe Signaltransduktionssysteme gehemmt wird.
Der interessante Zusammenhang zwischen schnellerer Entwicklung und dem Verlust der Fruchtkörperbildung könnte sich herausgebildet haben, um es M. xanthus zu ermöglichen, den Vorteil der Sporulation in Zellgruppen (die mindestens 72 Stunden in Anspruch nimmt) zugunsten einer beschleunigten Sporulation aufzugeben, um das Überleben unter einer Vielzahl von Einflüssen zu sichern, denen der Organismus in seinem natürlichen Lebensraum ausgesetzt ist.


