Forschungsbericht 2022 - Max-Planck-Institut für Dynamik komplexer technischer Systeme
Kontrolle und Regulation der Balance von Zellwachstum und Zellabbau
Control and regulation of balance of cell growth and degradation
Max-Planck-Institut für Dynamik komplexer technischer Systeme, Magdeburg
Einleitung
Computergestützte Simulationen sind mittlerweile in der Lage, die molekularen Grundlagen komplexer biochemische Vorgänge aufzuklären und somit therapeutische Ansätze zur Behandlung von Krankheiten zu unterstützen.
Die Methode der Molekulardynamiksimulationen basiert auf den physikalischen Fundamenten der Integration der Newtonschen Bewegungsgleichungen, so dass die Bewegungen aller Moleküle und Atome über die gesamte Simulationszeit verfolgt werden können. Um Systeme mit mehreren Hundertausenden oder Millionen von Teilchen zu betrachten, sind zeitlich aufwändige Simulationen auf Hochleistungsrechnern notwendig. Zurzeit sind durch Computersimulationen Zeitskalen von mehreren Mikrosekunden zugänglich.
Zusammenhang von Entzündungen und Krebs
Die Kontrolle und Regulation von Zellwachstum und Zellabbau ist für einen physiologischen und ausgeglichenen Stoffwechsel wichtig. Überwiegt einer von beiden führt das zu einem Ungleichgewicht mit dramatischen Auswirkungen auf den Organismus: chronische Entzündungen, Krebs und andere Krankheiten sind möglich. Chronische Entzündungen sind u.a. Triggerfaktoren für die Entstehung von malignen Tumoren. 2018 wurde der Nobelpreis in Medizin an James P. Allison und Rasuku Honjo verliehen für ihre bahnbrechende Forschung zur Wechselwirkung zwischen unkontrolliertem Zellwachstum und menschlicher Immunabwehr und deren Anwendung in der Krebstherapie.
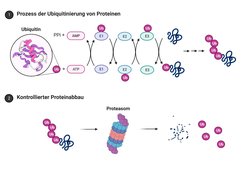
Die Ubiquitinierung ist ein komplexer biochemischer Prozess, in dem durch den Ablauf mehrere Enzyme (die Ubiquitin-Ligasen E1-E3) eine Markierung an ein Zielprotein angeheftet wird. Dieser Ubiquitinlabel ist selber ein kleines Protein und eine Markierung der Zielproteine. Diese Markierung ist ein Erkennungszeichen für den Abbau der so markierten Proteine im multi-protein Komplex Proteasom (siehe Abbildung 1). Die Stabilität eines Proteins ist eine zentrale Regulation des Metabolismus einer Zelle. 30% aller zellulären Proteine existieren nur für 10 Minuten, bevor sie wieder abgebaut werden. Die Gegenspieler der Ligasen sind die Enzyme Deubiquitinasen, die die Markierung durch Ubiquitinproteine wieder entfernen und Proteine recyceln und so ihren eigentlichen Aufgaben in der Immunabwehr oder in Signaltransduktionswegen wieder zuführen.
Der Mensch besitzt ungefähr 100 verschiedene derartige Deubiquitinasenenzyme, die z.T. Fähigkeiten zur unterschiedliche Erkennungen von markierten menschlichen Proteinen besitzen , die Schlüsselenzyme bestimmter Immunreaktionen sind. Bei Störung des Abbauprozesses kann so ungesteuertes Zellwachstum zur Entstehung von Tumoren führen oder die Immunabwehr gegen Bakterien und Viren supprimiert werden.
Die Untergruppe der OTUs wurden zuerst in Tumoren in Eierstöcken entdeckt und danach benannt (OTU = ovarian tumor deubiqutinases, Eierstocktumordeuibiquitnasen). Einige dieser OTUs weisen eine sehr hohe Selektivität hinsichtlich ihrer Erkennung und Funktion auf, andere dagegen scheinen viele Arten von ubiquitin-markierten Proteinen zu erkennen. Die strukturell verwandten OTUs OTUB1 und OTUB2 haben wichtige Funktionen bei der Regulierung von Infektionen und Autoimmunerkrankungen. Obwohl sie sich ähnlich sehen, ist ihre Funktionsweise doch unterschiedlich und konnte durch Molekulardynamiksimulationen beschrieben werden. [1]
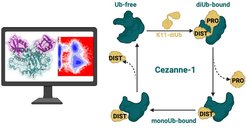
Die Deubiquitinase Cezanne-1 ist nicht nach dem französischen Maler benannt, sondern eine Abkürzung für die besondere Struktur und Funktion des Enzyms ‚cellular zinc finger anti-NF κB‘. Sie weist eine besondere Selektivität für eine bestimmte Verknüpfungsarte von Ubiquitinmolekülen auf, die bisher nicht vollständig verstanden ist. Cezanne-1 ist darin einzigartig und hebt sich von anderen OTUs ab. Obwohl es eine Reihe von Proteinstrukturen für Cezanne-1 gibt, können diese keine Einblicke in die Dynamik und die Flexibilität des Enzyms während seiner Reaktion geben. Das Enzym ist nicht starr, sondern verändert seine Struktur, wenn es das di-Ubiquitin Substrat zuerst erkennt, bindet und dann spaltet. [2] Durch aufwändige Computersimulationen ist es gelungen, die verschiedenen Zustände des Cezanne-1 Enzyms während seines Reaktionsmechanismus genau zu charakterisieren und dessen besondere Selektivität zu erklären. Derartige Informationen sind aus den statischen Proteinstrukturen nicht erhältlich, sondern erfordern die Analyse von einer großen Datenmenge aus den Computersimulationen mit modernen mathematischen Algorithmen.

Auch Bakterien und Viren kodieren DUB-ähnliche Aktivitäten, die die Ubiquitinationswege des Wirts stören sollen. [3] Bakterien, wie z.B. die Legionellen, besitzen eigene Deubiquitinaseenzyme, die die Immunabwehr des Menschen unterdrücken. [4] Das bakterielle Analog RavD des menschlichen OTULINs imitiert die Erkennung der bakteriellen Proteine durch das menschliche Immunsystem durch ähnliche Bindungseigenschaften. [5] Derartige Enzyme aus Pathogenen sind oft mit eukaryotischen DUBs entweder strukturell oder auch nur funktionell verwandt. Die Anwesenheit von ähnlichen imitierenden Enzymen in Viren z.B. im Human Papillomarivus (HPV) oder in RNA-Viren wie SARS-CoV und SARS-CoV2 führen zu einer verstärkten Produktion der Deubiquitinasen im Menschen, stören dessen Signaltransduktionswege und hemmen so die Immunabwehr.
Die Untersuchung der strukturellen Dynamik von regulierenden Proteinen kann zur Entwicklung neuer maßgeschneiderter Krebsmedikamente führen, die spezifischer sind und so weniger Nebenwirkungen aufweisen. [6], [7] Deubiquitinierende Enzyme sind seit kurzem auch als mögliche Ansätze für neue Therapeutika gegen Infektionen durch Bakterien und Viren erkannt worden.