Forschungsbericht 2019 - Max-Planck-Institute für biophysikalische Chemie
Neue Einblicke in die Struktur und Funktion des humanen Spleißosoms
Einführung
In jeder Zelle unseres Organismus gibt es eine Fülle vielgestaltiger Proteine, deren Baupläne in der Erbsubstanz DNA abgelegt sind. Um nach diesen Bauplänen Proteine herzustellen, wird ein Gen zunächst in die Rohfassung einer Boten-RNA (mRNA), die sogenannte prä-mRNA, umkopiert. In höheren Organismen liegt der Bauplan für ein Protein gewöhnlich nicht in einem Stück vor, sondern in mehreren Abschnitten, den Exons. Zwischen den Exons liegen Bereiche, die aus der prä-mRNA herausgeschnitten werden müssen – die Introns. Erst in diesem Schritt, prä-mRNA-Spleißen genannt, werden alle benötigten Exons lückenlos zu einer gebrauchsfertigen mRNA miteinander verbunden. Der Aufwand, der für diesen sehr kompliziert erscheinenden Prozess getrieben wird, lohnt sich, denn bei Bedarf können unterschiedliche Exons miteinander kombiniert und verknüpft werden. Damit liefert ein einzelnes Gen die Baupläne für verschiedene Proteine. Dieser als alternatives Spleißen bezeichnete Prozess erklärt, dass der Mensch mit nur gut 20.000 Genen ein Vielfaches an verschiedenen Proteinen herstellen kann.
Spleißosomen – molekulare Schneidetische
Der Ablauf der prä-mRNA-Spleißreaktion erfolgt in zwei Teilschritten über Phosphoester-Transferreaktionen (Abb. 1A). Diese werden durch gigantische molekulare Maschinen, Spleißosomen genannt, ausgeführt, die aus fünf kleinen RNA-Molekülen, den snRNAs U1, U2, U4, U5 und U6, und mehr als 150 Proteinen bestehen. Viele der Proteine sind in Subkomplexen vorformiert. So lagern sich zum Beispiel rund 50 spleißosomale Proteine mit den snRNAs zu RNA-Proteinpartikeln, den snRNPs (small nuclear ribonucleoproteins) U1, U2 sowie dem U4/U6.U5 tri-snRNP, zusammen. Die snRNPs übernehmen dabei ganz bestimmte Aufgaben (Abb. 1B). Überraschenderweise existieren Spleißosomen nicht als vorgefertigte Maschinen, sondern werden an jedem Intron aus den einzelnen Bausteinen neu zusammengesetzt. Im vollständigen Spleißosom, dem B Komplex, sind die Enden des Introns, die gespleißt werden sollen, bereits integriert, ihm fehlt aber noch ein aktives, katalytisches Zentrum.
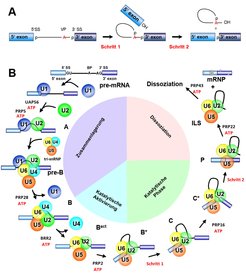
Erst nach drastischen Umlagerungen des B Komplexes entsteht ein katalytisch aktives Spleißosom, der sogenannte Bact Komplex, der die beiden Schritte der Spleißreaktion bewerkstelligt und nach Entlassen der mRNA wieder in seine Bausteine zerfällt (Abb. 1B). Weiterhin sind eine Reihe von Energie verbrauchenden Enzymen, sogenannte RNA-Helikasen, entscheidend an den zahlreichen, schrittweise erfolgenden Umlagerungen des Spleißosoms beteiligt (Abb. 1B). In den letzten Jahren ist es uns gelungen, unter Einsatz der Cryo-Elektronenmikroskopie (Cryo-EM) die 3D-Struktur der humanen spleißosomalen Komplexe der Aktivierungs- und katalytischen Phase aufzuklären. Diese Untersuchungen haben unser Verständnis der molekularen Architektur, der Funktion und der strukturellen Dynamik des Spleißosoms revolutioniert [1].
Die molekularen Scheren des Spleißosoms werden schrittweise aktiviert
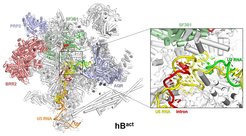
Die Cryo-EM-Strukturen des humanen U4/U6.U5 tri-snRNP-Partikels und der spleißosomalen B und Bact Komplexe haben die molekularen Mechanismen aufgedeckt, wie das Herzstück des katalytischen Zentrums, die U6 snRNA, schrittweise in eine katalytisch aktive Konformation im Bact Komplex gefaltet wird [1, 2, 5]. Dieses geschieht durch Verpaarung von U6 snRNA mit Sequenzabschnitten von U2 snRNA und durch Unterstützung zahlreicher Proteine, ein Prozess, der evolutionär hoch konserviert ist (Abb. 2).
RNA-Helikasen sind die Schrittmacher des Spleißosomenzyklus
Der Bact Komplex kann aber noch nicht den ersten Spleißschritt durchführen, da der Verzweigungspunkt (VP) des Introns (s. Abb. 1A) deutlich vom aktiven Zentrum entfernt durch ein Protein verankert ist. Dieses Protein muss zunächst durch die RNA-Helikase PRP2 entfernt werden. Die Kryo-EM-Struktur vom Bact Komplex legt einen neuartigen Mechanismus für die RNA-Helikase nahe: Sie dockt an das VP-bindende Protein an und löst dieses wahrscheinlich durch eine Konformationsänderung vom VP ab [2, 5]. Damit bekommt das Intronende mit dem VP genügend Beweglichkeit, kann am katalytischen Zentrum andocken und mit der 5'-Spleißstelle der prä-mRNA unter Ausbildung der Verzweigungsstruktur reagieren. Unsere Cryo-EM-Strukturen der sogenannten C und C* Komplexe zeigen, wie eine weitere RNA-Helikase (Prp16) die Verzweigungsstruktur des Introns über eine Drehbewegung aus dem katalytischen Zentrum des C-Komplexes entfernt, um dort für das Andocken der 3'-Spleißstelle der prä-mRNA Platz zu schaffen [3]. Dieser Umbau ist erforderlich, da das Spleißosom nur ein einziges katalytisches Zentrum hat, das für beide Spleißschritte benötigt wird.
Spleißosomen sind enorm dynamische molekulare Maschinen
Während unsere früheren biochemischen Untersuchungen die kompositionelle Dynamik des Spleißosoms deutlich gemacht hatten, haben die Cryo-EM-Strukturen der diversen spleißosomalen Komplexe jetzt offenbart, wie dramatisch die Dynamik der Strukturumlagerungen des Spleißosoms beim Übergang von einem Zustand zum nächsten sein kann. So können mehrere große Strukturelemente des Spleißosoms sukzessive Translokationsbewegungen von mehr als 20 Nanometern durchlaufen, ein Prinzip, das von anderen molekularen Maschinen wie beispielsweise dem Ribosom nicht bekannt ist.
Durch unsere Arbeiten sowie die unserer Kolleginnen und Kollegen, vor allem von Kiyoshi Nagai (LMB, Cambridge, UK) und Yigong Shi (Tsinghua Universität, Peking, China), ist ein lange gehegter Traum in wenigen Jahren in Erfüllung gegangen: dem Spleißosom bei der Arbeit zuschauen zu können. In Zukunft wird es unter anderem darum gehen, diejenigen Schaltprozesse, die alternativen Spleißvorgängen zugrunde liegen, auf struktureller Basis zu verstehen. Des Weiteren können wir jetzt untersuchen, wie sich bestimmte Mutationen in spleißosomalen Proteinen beziehungsweise snRNAs, die zu Krebs oder neurologischen Erkrankungen führen, auf die Spleißosom-Struktur auswirken. Dies kann zukünftig Ansatzpunkte zur Therapie dieser Krankheiten liefern.