Forschungsbericht 2013 - Max-Planck-Institut für terrestrische Mikrobiologie
Biochemie des mikrobiellen Methan-Zyklus
Einleitung
Methan ist der wahrscheinlich am häufigsten vorkommende Kohlenwasserstoff auf der Erde. Allein die Methanhydrat Lagerstätten an den marinen Kontinentalabhängen werden auf etwa 10.000 Gt Methan geschätzt. Methan wird vom Menschen als Brennstoff genutzt aber auch als Ausgangsrohstoff zur Erzeugung von Wasserstoff (H2) und für organische Synthesen. Methan kommt auch in der Atmosphäre vor, wo seine Konzentration sich in den letzten 200 Jahren verdoppelt hat, was von Besorgnis ist, da Methan ein Treibhausgas mit einem fast 25-fach höheren Treibhauspotential als CO2 ist. Die chemische oder mikrobielle Bildung von Methan aus H2 und CO2 wird als Möglichkeit diskutiert, den Energieträger Wasserstoff zu speichern [1].
Jährlich werden etwa 70 Gt Kohlenstoff aus CO2 durch Photosynthese in pflanzlicher Biomasse netto fixiert (Abb. 1), wovon etwa 60 Gt auf terrestrische Biomasse und 10 Gt auf marine Biomasse entfallen. Von der Biomasse, von der der größte Teil aus Lignozellulosen besteht, wird der Löwenanteil durch Mikroorganismen und Tiere wieder in CO2 veratmet (remineralisiert). Aber etwa 1% gelangt in Biotope, in denen es weder Sauerstoff noch andere positive Elektronenakzeptoren wie Nitrat, Nitrit, Fe(III), Mn(IV) oder Sulfat gibt, so in Süßwassersedimenten und Sümpfen, im Intestinaltrakt von Mensch und Tier im Allgemeinen und im Pansen von Wiederkäuern im Besonderen, aber auch in Sulfat-verarmten marinen Sedimenten. In diesen Biotopen wird die Biomasse, vorwiegend deren verbleibende Zellulose-Bestandteile, zunächst unter Beteiligung von anaeroben Protozoen, Pilzen und bzw. oder Bakterien hauptsächlich zu Essigsäure, CO2 und H2 fermentiert, die dann von methanogenen Archaea zu Methan umgesetzt werden. Im Intestinaltrakt beschränkt sich die Methanbildung auf H2 und CO2 als Substrate, da die Essigsäure vom Tier resorbiert und damit dem Zugriff der methanogenen Archaea entzogen wird. Global addiert sich die jährlich durch methanogene Archaea gebildete Methanmenge auf 1 Gt [2]. Dazu kommt eine kleinere Menge Methan, die in den Weltmeeren durch aereobe Bakterien aus Methylphosphonat gebildet wird, der Bestandteil der Zellwand von marinen Thaumarchaeota ist [3]. Eine Gt Methan hat einen Brennwert von 50 EJ (E [Exa] = 1018). Zum Vergleich: Der globale Primärenergieverbrauch beträgt derzeit rund 500 EJ.
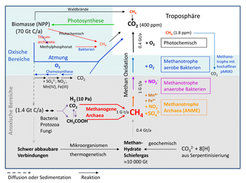
Abb. 1: Schematische Darstellung des Methan-Zyklus. NPP, Nettoprimärproduktion. Genaue Erläuterung im Text.
Methan entsteht auch thermochemisch aus Biomasse bei der Kohlebildung, im reduzierenden Teil der Flamme bei der Verbrennung von Biomasse, photochemisch aus Verbindungen wie Pektin und rein anorganisch durch Reduktion von CO2 mit Fe(II) während Serpentinisierungs-prozessen (Abb. 1).
Bis vor wenigen Jahren wurde allgemein angenommen, dass Methan nur in aeroben Prozessen biologisch abgebaut werden kann. Alle bekannten methanotrophen Mikroorganismen waren auf O2 angewiesen. Inzwischen steht aber fest, dass Methan auch in Biotopen ohne Licht und Sauerstoff zu CO2 oxidiert wird, wobei Nitrat, Nitrit, Fe(III), Mn(IV) und Sulfat als Elektronenakzeptoren dienen [4]. Methan in der Atmosphäre wird zum allergrößten Teil photochemisch remineralisiert, nur ein kleiner Teil gelangt von der Atmosphäre zurück in Böden und Gewässer und wird dort durch aerobe Methanotrophe mit hoher Methan-Affinität zu CO2 umgesetzt ([5]; Abb. 1).
Anaerobe Bildung und Oxidation von Methan werden von dem gleichen Enzymsystem katalysiert
Eine der ersten aufregenden Entdeckungen bei der Untersuchung der Biochemie der Methanbildung war, dass der eigentliche Methan-bildende Schritt in methanogenen Archaea von einem Enzym katalysiert wird, dessen prosthetische Gruppe ein Nickeltetrapyrrol ist, das im Ni(I) Zustand vorliegen muss, um aktiv zu sein (Abb. 2). Das Enzym katalysiert die Reduktion von Methyl-Coenzym M (CH3-S-CoM) mit Coenzym B (HS-CoB) zu Methan und dem Heterodisulfid CoM-S-S-CoB (Reaktion (1), s. auch Abb. 3).
(1) CH3-S-CoM + HS-CoB ⇌ CH4 + CoM-S-S-CoB ∆Go = -30 kJ/mol
Methyl-Coenzym M Reduktase (McrABG), deren Kristallstruktur mit einer Auflösung von 1,16 Å von uns bereits 1997 ermittelt wurde, kommt in allen methanogenen Archaea vor [6]. Sie ist aus den Untereinheiten McrA, McrB und McrG aufgebaut, von denen die McrA-Untereinheit fünf hoch konservierte post-translationale Modifikationen enthält, darunter ein Thioglycin, deren Funktionen bisher noch nicht aufgeklärt werden konnten.
![Abb. 2: Struktur von Kofaktor F430 aus methanogenen und methanotrophnen Archaea. Die beiden grau hinterlegten Methylgruppen stammen aus S-Adenosylmethionin [6]. ANME steht für methanotrophe Archaea, die zusammen mit Sulfat-reduzierenden Bakterien die anaerobe Oxidation von Methan katalysieren.](/11602187/original-1508157043.jpg?t=eyJ3aWR0aCI6MjQ2LCJvYmpfaWQiOjExNjAyMTg3fQ%3D%3D--951e8b042538e29094e7f9fef90d89e7593da585)
Abb. 2: Struktur von Kofaktor F430 aus methanogenen und methanotrophnen Archaea. Die beiden grau hinterlegten Methylgruppen stammen aus S-Adenosylmethionin [6]. ANME steht für methanotrophe Archaea, die zusammen mit Sulfat-reduzierenden Bakterien die anaerobe Oxidation von Methan katalysieren.
McrA enthält nicht nur die fünf post-translationalen Modifikationen, sondern auch hoch konservierte Abschnitte, die als Gensonden für die Identifizierung von methanogenen Archaea Verwendung finden. Mithilfe dieser Gensonden sind kürzlich zwei neue Ordnungen von Methanbildnern entdeckt worden, nämlich die Methanocellales und die Methanoplasmatales. Dank derselben Gensonden war bereits vor zehn Jahren entdeckt worden, dass methanotrophe Archaea (ANME-1, ANME-2 und ANME-3), die an der anaeroben Oxidation von Methan mit Sulfat beteiligt sind, mcr-Gene enthalten, die offensichtlich auch exprimiert werden: Methanotrophe Archaea enthalten hohe Konzentrationen an Mcr Protein. Aber es gelang erst in den letzten Jahren nachzuweisen, dass McrABG aus methanotrophen Archaea auch Methyl-Coenzym M und Coenzym B als Substrate verwenden und dass - so zeigten Laborversuche - Methyl-Coenzym M Reduktase auch tatsächlich die Oxidation von Methan katalysieren kann, und zwar mit spezifischen Raten, die denen in vivo entsprechen. Letzteres war entscheidend, denn auf Grund des ∆Go von -30 kJ/mol wurde zunächst allgemein angenommen, dass Reaktion (1) irreversibel verläuft [7].
Bemerkenswert ist, dass Methyl-Coenzym M Reduktase aus ANME-1 Archaea einen modifizierten Kofaktor F430 enthält (Abb. 2). In Methyl-Coenzym M Reduktase aus ANME-2 und ANME-3 Archaea und in allen methanogenen Archaea wird nur der unmodifizierte F430 Kofaktor gefunden. Eine funktionelle Erklärung steht dafür noch aus.
Inzwischen scheint es, dass auch die anaerobe Oxidation von Methan mit Nitrat, Fe(III) und Mn(IV) in methanotrophen Archaea unter Beteiligung von Methyl-Coenzym M Reduktase abläuft [4]. Da sowohl methanogene als auch methanotrophe Archaea Methyl-Coenzym M Reduktase in hohen Konzentrationen - bis zu 10% ihres löslichen Zellproteins - enthalten, dürfte das Nickelenzym eines der mengenmäßig häufigsten Enzyme in der anaeroben Welt sein.
Bezüglich des Katalysemechanismus gibt es in der Literatur unterschiedliche Ansichten, insbesondere über die Startreaktion, wobei keiner der Mechanismen eine Erklärung für die posttranslationalen Modifikationen gibt, die auch in Methyl-Coenzym M Reduktasen von methanotrophen Archaea gefunden werden. Mechanismus 1, für den es die meisten experimentellen Evidenzen gibt, geht davon aus, dass CH3-S-CoM im aktiven Zentrum mit Ni(I) von F430 zu Ni(III)-CH3 reagiert. Er berücksichtigt aber nicht, dass die geforderte nukleophile Substitution thermodynamisch äußerst ungünstig zu sein scheint, was theoretische Rechnungen ergeben haben. Dagegen behauptet Mechanismus 2, dass Ni(I) von F430 im ersten Schritt ein Elektron an den Schwefel von CH3-S-CoM abgibt, wobei ein Methyl-Radikal entsteht, was thermodynamisch ohne Problem zu sein scheint. Aus Messungen von Isotopeneffekten, die den Rahmen für den Katalysemechanismus abstecken, geht jedoch hervor, dass noch vieles unverstanden ist [8].
Abschließend sei erwähnt, dass die anaerobe Oxidation von Methan mit Nitrit nicht durch Archaea, sondern durch Bakterien katalysiert wird, die 2 NO in N2 und O2 dismutieren und so Sauerstoff für die Oxidation von Methan bereitstellen, die durch eine Membran-gebundene Methan-Monooxygenase katalysiert wird [9].
Energetische Kopplungen durch Flavin-basierte Elektronen-Bifurkationen
Die am Methan-Zyklus beteiligten anaeroben Mikroorganismen benötigen fast alle Ferredoxin für Reduktionsreaktionen, wobei das Problem auftaucht, dass das Redoxpotenzial von Ferredoxin (-450 mV) negativer ist als das der meisten physiologischen Elektronendonatoren. So beträgt das Redoxpotenzial von H2 bei pH 7 und einem physiologischen H2 Partialdruck von 10 Pa nur etwa -300 mV. Wie kann Ferredoxin (-450 mV), das zum Beispiel für die Reduktion von CO2 im ersten Schritt der Methanogenese benötigt wird, durch H2 (-300 mV) reduziert werden, obwohl diese Reaktion doch endergon ist (Abb. 3)? Diese Frage war bis vor ein paar Jahren offen [10].
![Abb. 3: Schema des Stoffwechsels von methanogenen Archaea, die auf H2 und CO2 als Energiequelle wachsen. Im Stoffwechsel wird die endergone Reduktion von Ferredoxin(-450 mV) mit H2 (-300 mV) mit der exergonen Reduktion von CoM-S-S-CoB (-100 mV) mit H2 (-300 mV) durch Flavin-basierte Elektronen-Bifurkation gekoppelt. MFR, Methanofuran; H4MPT, Tetrahydromethanopterin; F420, Coenzym F420; CoM-S-S-CoB, Heterodisulfid aus Coenzym M und Coenzym B [2, 10].](/11602195/original-1508157043.jpg?t=eyJ3aWR0aCI6MjQ2LCJvYmpfaWQiOjExNjAyMTk1fQ%3D%3D--ad1b518202ffc3b6e28592f134fa1b15bfaf4876)
Abb. 3: Schema des Stoffwechsels von methanogenen Archaea, die auf H2 und CO2 als Energiequelle wachsen. Im Stoffwechsel wird die endergone Reduktion von Ferredoxin(-450 mV) mit H2 (-300 mV) mit der exergonen Reduktion von CoM-S-S-CoB (-100 mV) mit H2 (-300 mV) durch Flavin-basierte Elektronen-Bifurkation gekoppelt. MFR, Methanofuran; H4MPT, Tetrahydromethanopterin; F420, Coenzym F420; CoM-S-S-CoB, Heterodisulfid aus Coenzym M und Coenzym B [2, 10].
Schlüssel zum Verständnis war die Entdeckung, dass in am Methan-Zyklus beteiligten Buttersäure-bildenden Clostridien die endergone Reduktion von Ferredoxin (-450 mV) mit NADH (-280 mV) durch Kopplung mit der exergonen Reduktion von Crotonyl-CoA (-30 mV) mit NADH zu Butyryl-CoA möglich wird. Diese gekoppelte Reaktion wird von einem cytoplasmatischen Enzymkomplex Bcd/EtfAB katalysiert, der als prosthetische Gruppen nur FAD enthält (Abb. 4, zweite Zeile). In Analogie zur Rolle von Ubichinon in der Atmungskette wurde postuliert, dass durch ein-Elektronen Oxidation des mit zwei Elektronen reduzierten FAD ein Flavin-Radikal entsteht, dessen Redoxpotenzial dem von Ferredoxinen entspricht: Also eine oxidationsgetriebene Reduktion, deren Elektronen-Bifurkation Flavin basiert ist [10].
![Abb. 4 Cytoplasmatische Enzymkomplexe mit Flavin-basierter Elektronen-Bifurkation. Bisher sind vier nicht-homologe Enzymfamilien entdeckt worden [10].](/11602243/original-1508157045.jpg?t=eyJ3aWR0aCI6MjQ2LCJvYmpfaWQiOjExNjAyMjQzfQ%3D%3D--a7efc0f59b1686b0637bcc5699a75b36eb90db50)
Es hat dann nicht lange gedauert, das Problem der endergonen Reduktion von Ferredoxin (-450 mV) mit H2 (-300 mV) in methanogenen Archaea zu lösen (Abb. 3). Die Reduktion wird möglich durch Kopplung mit der Reduktion von CoM-S-S-CoB - Reaktion (1) (-100 mV) - mit H2 (-300 mV). Die beiden gekoppelten Reaktionen werden von einem cytoplasmatischen Enzymkomplex MvhAGD/HdrABC katalysiert, der gebunden an HdrA ein FAD enthält (Abb. 4, erste Zeile).
Schlussfolgerung
Soweit wir es heute bereits übersehen, spielt die Flavin-basierte Elektronen-Bifurkation im Stoffwechsel der meisten anaeroben Mikroorganismen eine zentrale Rolle (Abb. 4). Kaum zu glauben, dass vermeintlich längst aufgeklärte Stoffwechselwege noch so viele neue Entdeckungen bergen. Ehe diese gehoben sind, wird es wenig sinnvoll sein, zu versuchen, über systembiologische Analysen und synthetische Ansätze zu Mikroorganismen mit verbesserten Stoffwechselleistungen zu gelangen, wie das ja auch bisher weitgehend erfolglos versucht worden ist.
Kollaborationen
Die hier vorgestellten Ergebnisse sind in Zusammenarbeit entstanden mit Seigo Shima an unserem Institut, mit Wolfgang Buckel von der Philipps-Universität Marburg, mit Ulrich Ermler vom Max-Planck-Institut für Biophysik in Frankfurt/Main und mit Bernhard Jaun von der Eidgenössisch-Technischen Hochschule (ETH) Zürich.
Literaturhinweise
DOI: 10.1146/annurev.biochem.030508.152103
DOI: 10.1038/nrmicro1931
DOI: 10.1126/science.1219875
DOI:10.1038/nature12375
DOI: 10.1111/j.1758-2229.2009.00038.x
DOI: 10.1099/00221287-144-9-2377
DOI:10.1038/nature09015
DOI: 10.1021/ja406485z
DOI:10.1042/BST0390243
DOI: 10.1016/bbabio.2012.07.002

![Abb. 2: Struktur von Kofaktor F430 aus methanogenen und methanotrophnen Archaea. Die beiden grau hinterlegten Methylgruppen stammen aus S-Adenosylmethionin [6]. ANME steht für methanotrophe Archaea, die zusammen mit Sulfat-reduzierenden Bakterien die anaerobe Oxidation von Methan katalysieren. Abb. 2: Struktur von Kofaktor F430 aus methanogenen und methanotrophnen Archaea. Die beiden grau hinterlegten Methylgruppen stammen aus S-Adenosylmethionin [6]. ANME steht für methanotrophe Archaea, die zusammen mit Sulfat-reduzierenden Bakterien die anaerobe Oxidation von Methan katalysieren.](/11602187/original-1508157043.jpg?t=eyJ3aWR0aCI6ODQ4LCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTE2MDIxODd9--1da9aaab091ce3002dfe2c3244cab8b78664491b)
![Abb. 3: Schema des Stoffwechsels von methanogenen Archaea, die auf H2 und CO2 als Energiequelle wachsen. Im Stoffwechsel wird die endergone Reduktion von Ferredoxin(-450 mV) mit H2 (-300 mV) mit der exergonen Reduktion von CoM-S-S-CoB (-100 mV) mit H2 (-300 mV) durch Flavin-basierte Elektronen-Bifurkation gekoppelt. MFR, Methanofuran; H4MPT, Tetrahydromethanopterin; F420, Coenzym F420; CoM-S-S-CoB, Heterodisulfid aus Coenzym M und Coenzym B [2, 10]. Abb. 3: Schema des Stoffwechsels von methanogenen Archaea, die auf H2 und CO2 als Energiequelle wachsen. Im Stoffwechsel wird die endergone Reduktion von Ferredoxin(-450 mV) mit H2 (-300 mV) mit der exergonen Reduktion von CoM-S-S-CoB (-100 mV) mit H2 (-300 mV) durch Flavin-basierte Elektronen-Bifurkation gekoppelt. MFR, Methanofuran; H4MPT, Tetrahydromethanopterin; F420, Coenzym F420; CoM-S-S-CoB, Heterodisulfid aus Coenzym M und Coenzym B [2, 10].](/11602195/original-1508157043.jpg?t=eyJ3aWR0aCI6ODQ4LCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTE2MDIxOTV9--0fc9894aaf99477a828cd87447c7ff70913fae2a)
![Abb. 4 Cytoplasmatische Enzymkomplexe mit Flavin-basierter Elektronen-Bifurkation. Bisher sind vier nicht-homologe Enzymfamilien entdeckt worden [10]. Abb. 4 Cytoplasmatische Enzymkomplexe mit Flavin-basierter Elektronen-Bifurkation. Bisher sind vier nicht-homologe Enzymfamilien entdeckt worden [10].](/11602243/original-1508157045.jpg?t=eyJ3aWR0aCI6ODQ4LCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTE2MDIyNDN9--c20cdcb0e6cd28850d84335561c0770a22418bf2)