Forschungsbericht 2013 - Max-Planck-Institut für molekulare Genetik
Lange, nicht-kodierende RNAs als Regulatoren der Transkription beim Menschen
Long non-coding RNAs as regulators of transcription in human
Einführung
Lange, nicht-kodierende RNAs (ncRNAs) sind große Ribonukleinsäuremoleküle, die nicht in Proteine übersetzt werden, sondern als RNA-Transkripte eine Funktion im Organismus übernehmen. Ihre Entdeckung und die schrittweise Aufklärung ihrer Funktion führt zu einem Umdenken in der modernen Molekularbiologie. Für die meisten langen ncRNAs ist deren Funktion in der Zelle noch weitgehend ungeklärt. Es gibt allerdings Hinweise, dass sie die Wirkungsweise von Transkriptionsfaktoren modulieren können. Dabei können sie sowohl als Ko-Faktoren agieren und die Aktivität von Transkriptionsfaktoren direkt beeinflussen als auch die Assoziierung und Aktivität weiterer Ko-Regulatoren beeinflussen.
Untersuchungen des Transkriptoms, also derjenigen Gene, die in Ribonukleinsäure umgeschrieben werden, haben in Verbindung mit den jüngsten Entwicklungen der RNA- und DNA-Sequenzierungstechnologien eine nahezu unendliche Menge an Daten über zahlreiche Säugetier- und menschliche Zelllinien und Gewebe bereitgestellt. Zusammen mit aktuellen Erkenntnissen über das Bindungsverhalten von Transkriptionsfaktoren, die Modifikation von chemischen Gruppen an Histonen und die Konformation von Chromatin bilden diese Daten die Basis für die Arbeit der Forschungsgruppe im Labor.
Enhancer
Enhancer sind regulatorische Sequenzen im Genom, an die spezifische Proteine, die sogenannten Transkriptionsfaktoren, binden und dadurch die Transkription, also Abschrift von Genen oder deren Translation, also Übersetzung in Proteine, verstärken oder unterdrücken. Im Gegensatz zu anderen Transkriptionsfaktor-Bindungsstellen, wie zum Beispiel den Promotoren, stehen die enhancer in keiner definierten räumlichen Beziehung zu den von ihnen regulierten Genen. Sie können sich in deren Nähe, aber auch mehrere Millionen Basen vor oder hinter demjenigen Gen befinden, dessen Aktivität sie regulieren. Enhancer wurden bereits 1981 entdeckt und als DNA-abhängige Regulatoren der Transkription beschrieben. Die molekularen Mechanismen, nach denen sie funktionieren, sind aber noch immer umstritten [1].
Mithilfe von analytischen und bioinformatischen Methoden, die speziell für diese Fragestellung entwickelt wurden, konnten in den letzten Jahren zahlreiche enhancer vorhergesagt und identifiziert werden. Dank aktueller Untersuchungen, die sich auf Chromatin-Markierungen, ChIP-sequencing-Experimente und Hochdurchsatz-Sequenzierungen stützen, wächst auch das Wissen über die allgemeinen Eigenschaften der enhancer im gesamten Genom. Dies ist die Grundlage für weitere Untersuchungen an enhancern, langen ncRNAs und weiteren assoziierten Faktoren, die an der Genregulation beteiligt sind.
Lange, nicht-kodierende RNAs und Enhancer
Die Forschungsgruppe „Lange nicht-kodierende RNAs“ am Max-Planck-Institut für molekulare Genetik untersucht die Funktion dieser speziellen RNAs auf molekularem Niveau. Ein Hauptaugenmerk der Wissenschaftler richtet sich auf die Bedeutung der langen ncRNAs für die Funktion von enhancern. Dieses Zusammenspiel stellt sich als zunehmend wichtiger heraus, und von der Aufklärung der Rolle der langen ncRNAs bei der enhancer-Aktivität versprechen sich die Forscher neue Erkenntnisse über die eigentliche, genaue Funktionsweise von enhancern [2, 3].
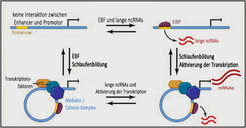
In zahlreichen Untersuchungen konnten Ørom und andere Wissenschaftler zeigen, dass lange ncRNAs die Expression verschiedener Gene gezielt regulieren. Sowohl aktivierende als auch reprimierende Funktionen der langen ncRNAs wurden dabei gefunden. Weiterhin spielen die ncRNAs eine entscheidende Rolle während der Differenzierung von embryonalen Stammzellen in der Maus. Gemeinsam mit Kooperationspartnern des Wistar Instituts in Philadelphia konnten Ørom und seine Mitarbeiter zeigen, dass bei dieser enhancer-ähnlichen Funktion noch weitere Proteinkomplexe beteiligt sind, die für die Initialisierung der enhancer-Promotor-Interaktionen eine Rolle spielen (Abb. 1) [4].
Bidirektionale Promotoren
Vor kurzem wurde in verschiedenen Studien über die Transkription von nicht-kodierenden RNAs im Bereich der Promotoren von sehr aktiven Genen berichtet. Jüngste Untersuchungen bestätigen das genomweite Vorkommen von bidirektionalen Promotoren und charakterisieren einige Eigenschaften dieser Gruppe von langen ncRNAs. Die Berliner Wissenschaftler vermuten, dass bidirektionale Promotoren die Genexpression in beide Leserichtungen des DNA-Doppelstranges regulieren können (Abb. 2). Dabei bedienen sie sich der gleichen Gruppe von Transkriptionsfaktoren. Dies ermöglicht es den Wissenschaftlern, die ko-regulierte Expression sowohl von langen ncRNAs als auch von protein-kodierenden Genen an einer spezifischen Stelle innerhalb des DNA-Doppelstranges zu untersuchen. Ihr Ziel ist es herausfinden, aus welchem Grund lange ncRNAs an bidirektionalen Promotoren exprimiert werden: Regulieren die langen ncRNAs direkt die Transkription oder Translation des gegenläufigen Protein-kodierenden Gens oder erfüllen sie möglicherweise eine Funktion in spezifischen Signalwegen und agieren dabei komplementär zu den Protein-kodierenden Genen, mit denen sie bidirektional transkribiert werden?
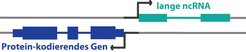
In menschlichen embryonalen Stammzellen konnte auf dem gegenläufigen DNA-Strang eines bidirektionalen Promotors im Bereich von bis zu 2000 Basenpaaren von der Startsequenz des Protein-kodierenden Gens entfernt die Expression von über 2000 langen ncRNAs nachgewiesen werden [5]. Ørom und seine Kollegen weisen darauf hin, dass 85% aller Promotoren von Protein-kodierenden Genen bidirektional transkribiert werden. Durch die Untersuchung von Promotoren, an denen lange ncRNAs und Protein-kodierende Gene paarweise exprimiert und ko-transkriptionell reguliert werden, versprechen sich die Forscher weitere Einblicke in die Mechanismen, über die lange ncRNAs Protein-kodierende Gene regulieren.
Bindung nicht-kodierender RNA an den Mediator-Komplex
Ein weiteres aktuelles Projekt der Forscher befasst sich mit dem Mediator-Komplex. Dies ist ein großer Komplex aus vielen Proteinen, der eine essenzielle Rolle bei der Vermittlung der Bindung zwischen enhancer and Promotor spielt [6]. Vor kurzem konnte gezeigt werden, dass der Mediator-Komplex direkt mit langen ncRNAs interagiert und über einen RNA-Protein-Komplex eine von diesem Gefüge abhängige Chromatinschlaufe herbeiführt [7]. Bei Patienten mit geistiger Behinderung wurde eine Reihe von Mutationen des Mediator-Komplexes nachgewiesen. Werden diese Mutationen in die Untereinheiten eines intakten Mediator-Komplexes eingefügt, verliert dieser seine Funktion und die Fähigkeit zur Bindung von RNA-Molekülen. Durch die Aufklärung der molekularen Mechanismen, die dieser Regulation zugrunde liegen, erhoffen sich Ørom und seine Kollegen neue Erkenntnisse darüber, wie Veränderungen einzelner Aminosäuren, die die Bindung von regulatorischen RNAs beeinträchtigen, zu Erkrankungen beim Menschen führen können.
Ausblick
Das besondere Interesse der Forschungsgruppe gilt den erst vor kurzem entdeckten langen ncRNAs. Für ihre Untersuchungen nutzen die Forscher Transkriptions-Assays, Experimente zur RNA-Protein-Interaktion und auch bioinformatorische Ansätze, um regulatorische Abschnitte im Genom zu identifizieren. Mithilfe neuer Experimente wollen sie herausfinden, wie RNAs die Genexpression regulieren. Das nächste Ziel der Gruppe ist es, die Wirkungsweise von enhancern und die Rolle der langen ncRNAs an diesen Regulationsmechanismen der Genexpression aufzuklären. Durch RNA-Sequenzierung und weitere, umfangreich angelegte Experimente erhoffen sich die Forscher allgemeingültige Informationen über die Regulationsmechanismen von langen ncRNAs. In Zusammenarbeit mit anderen Gruppen des Max-Planck-Instituts für molekulare Genetik sind Untersuchungen an Mäusen geplant, um das molekulare Wechselspiel zwischen Transkriptionsfaktoren und langen ncRNAs im lebenden Organismus zu studieren.

