Forschungsbericht 2003 - Max-Planck-Institut für Psychiatrie
Proteinmarker für psychiatrische und neurologische Krankheiten
Proteomics und Biomarker (Prof. Dr. Christoph Turck)
MPI für Psychiatrie, München
Um die komplexen pathophysiologischen Prozesse bei der Entstehung von Krankheiten im Detail zu verstehen, genügt es nicht, nur das Genom eines Organismus zu kennen. Die Mehrzahl aller Krankheiten wird sowohl durch genetische als auch durch Umwelteinflüsse hervorgerufen, ist also multifaktoriell bedingt. Daher ist ein umfassender Ansatz zur Identifizierung so genannter Marker erforderlich: körpereigener Substanzen, welche die pathologisch veränderten Funktionen des jeweiligen Krankheitsprozesses widerspiegeln. Für die aktuelle medizinische Forschung bedeutet dies, dass nach Markern nicht nur auf der Ebene der Nukleinsäuren, DNA und RNA, sondern auch der Proteine gesucht werden muss.
Als die eigentlichen Funktionsträger der Zelle repräsentieren die Proteine den Phänotyp eines Organismus - sein Erscheinungsbild - am besten. Die Gesamtheit aller Proteine eines Organismus wird heute analog zum Genom, welches die Gesamtheit der Gene enthält, das Proteom genannt. Es ist im Gegensatz zum Genom ein äußerst dynamisches System, dessen Zusammensetzung sich nicht nur in den verschiedenen Geweben eines Organismus unterscheidet, sondern sich auch im zeitlichen Verlauf ständig verändert. Daher liegt es nahe, die Unterschiede der Proteome zwischen gesunden und kranken Geweben zur Charakterisierung von Krankheiten zu verwenden.
Infolge der Strukturaufklärung (Sequenzierung) des menschlichen Genoms steht inzwischen fest, dass sich die Anzahl der Gene des Menschen nicht wesentlich von der weniger komplexer Lebensformen unterscheidet. Das wirft die Frage auf, warum der Mensch dennoch eine ungleich höher entwickelte Lebensform darstellt als alle anderen Organismen der Tier- und Pflanzenwelt. Die Komplexität wird offensichtlich nicht durch die Anzahl der Gene eines Organismus bestimmt, sondern vor allem durch Mechanismen, die zu Modifikationen der als Funktionsträger dienenden Proteine führen (Abb. 1).
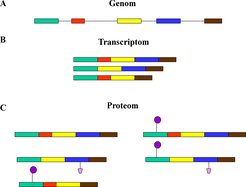
Das Verständnis der biologischen Synthese, Struktur und Funktion von Proteinen bildet daher auch den Schlüssel zur Untersuchung von Krankheitsmechanismen. Aufgrund dieser zentralen Bedeutung überrascht es nicht, dass derzeit sowohl im akademischen wie auch im industriellen Bereich große Anstrengungen unternommen werden, um Marker für Krankheiten auf der Proteinebene zu gewinnen. Das Endziel der Proteomanalyse in der Medizin ist die Aufklärung der quantitativen und qualitativen Veränderungen der Proteine in den Geweben und Körperflüssigkeiten bei gesunden und kranken Individuen. Dazu ist unter anderem die Bestimmung der Interaktionen der Proteine, ihrer Menge, Dynamik und ihrer Modifikationen sowie ihrer Lokalisierung in der Zelle erforderlich. Die identifizierten Markerproteine können dann im klinisch-chemischen Routinelabor zu diagnostischen Zwecken verwendet werden.
Diese Analyse von Proteinen ist wesentlich komplizierter als die der Nukleinsäuren und erfordert eine Reihe von Methoden und Großgeräten. Die klinische Proteomik stützt sich zunächst auf Körperflüssigkeiten wie Blut, Urin oder Liquor, die Flüssigkeit, die das zentrale Nervensystem umgibt. Die Proben können sowohl zur Krankheitsdiagnose als auch zur Überprüfung von Therapieansätzen benutzt werden. Neben universell verbreiteten Proteinen, wie Albumin und Immunoglobulinen, kommen in Körperflüssigkeiten viele Proteine vor, die aus bestimmten Geweben stammen. Allerdings sind diese nur in sehr geringen Mengen vorhanden, weshalb ihre Analyse extrem empfindliche Methoden erfordert.
Außerdem wird die Proteomanalyse durch die Tatsache erschwert, dass es bei menschlichen Proben infolge der genetischen Variationen zu individuellen Unterschieden in der Proteinzusammensetzung kommt. Weitere Variationen werden durch Umwelteinflüsse hervorgerufen. Dies gestaltet die Auffindung von zuverlässigen Krankheitsmarkern beim Menschen wesentlich komplizierter als bei Tiermodellen, die durch einen homogenen genetischen Hintergrund und eine experimentell kontrollierbare Umwelt gekennzeichnet sind.
Projekte der Proteomik
Im Folgenden werden Projekte vorgestellt, welche die Arbeitsgruppe "Molecular, Cellular, Clinical Proteomics" (Chris Turck) in Zusammenarbeit mit anderen Gruppen am Max-Planck-Institut für Psychiatrie mithilfe der Proteomanalyse bearbeitet. Es handelt sich um Untersuchungen der Signalweiterleitung in definierten zellulären Systemen, Studien von Tiermodellen für Depression, Angst und multiple Sklerose sowie die Evaluierung von klinischem Untersuchungsmaterial.
Zur Identifizierung geeigneter Marker wird das Proteom anhand von Liquorproben analysiert. Die Gewinnung und Aufbereitung des Liquors für Zwecke der Proteomik erfordert spezielle klinische und Labormethoden, die die Arbeitsgruppe "Pharmakokinetik und Liquoranalytik" (Manfred Uhr) entwickelt. Zur Fraktionierung der Proteingemische sind ebenfalls besondere Verfahren erforderlich, hauptsächlich die zweidimensionale Elektrophorese und die multidimensionale Chromatographie. Im letzten Schritt der Analyse ziehen die Forscher um Chris Turck die Massenspektrometrie zur Identifizierung der Proteine heran (Abb. 2). Die Kombination dieser Technologien ermöglicht nicht nur die Trennung einer großen Anzahl von Proteinen aus dem komplexen Liquorgemisch, sondern auch ihre exakte Quantifizierung. Nachdem auf diese Weise bereits mehr als 400 Proteine im Liquor bestimmt werden konnten, suchen die Wissenschaftler jetzt nach charakteristischen Markern sowohl für Depression und Angst als auch für Multiple Sklerose.

Bei einer Reihe von psychiatrischen und neurologischen Krankheiten, vor allem bei der Depression, spielt die Störung der Stresshormonregulation eine bedeutende Rolle. Eine solche Störung liegt beispielsweise bei der zellulären so genannten Glukokortikoidresistenz vor. Um hierfür die molekularen Ursachen zu erforschen, stellte die Arbeitsgruppe "Chaperone" (Theo Rein) durch Klonierung und Zufallsmutagenese Zellen her, deren Glukokortikoid-Signaltransduktion gestört ist. Um die Unterschiede zwischen den ursprünglichen Zellen mit intakter Signalweiterleitung und den mutierten Zellen zu beschreiben, identifizierten die Forscher um Theo Rhein und Chris Turck unterschiedlich exprimierte Proteine mittels zweidimensionaler Elektrophorese und Massenspektrometrie. Die gefundenen Regulatoren steuern die Aktivität der Glukokortikoide und könnten neue Behandlungsstrategien für psychiatrische Störungen ermöglichen.
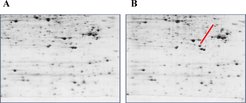
In Zusammenarbeit mit der Arbeitsgruppe "Verhaltensneuroendokrinologie" (Rainer Landgraf) führen die Wissenschaftler um Chris Turck Proteomanalysen von Gehirnarealen bei gezüchteten Mauslinien durch, die sich durch erhöhtes oder erniedrigtes Angstverhalten auszeichnen. Die Analyse der Proteinextrakte mittels zweidimensionaler Gelelektrophorese ergab einen signifikanten Unterschied in der Expression eines Proteins, das ein wichtiges Enzym für den Metabolismus dieser Neuronen darstellt (Abb. 3). Nach der Identifizierung dieses Markers für den Ausprägungsgrad von Angstverhalten in den gezüchteten Mauslinien entwickelten die Forscher mithilfe eines Antikörpers einen Hochdurchsatz-Assay zum Nachweis in Blutproben von Patienten. Die Ergebnisse des Screenings werden zeigen, ob ein solcher Test zur Diagnose von Angsterkrankungen eingesetzt werden kann.
Im neurologischen Bereich untersucht Turcks Team anhand eines Tiermodells für Multiple Sklerose (Experimental Allergic Encephalomyelitis) ebenfalls Gehirn- und Rückenmarksgewebe auf spezifische Markerproteine. Für dieses Projekt arbeiten die Wissenschaftler mit der Max-Planck-Arbeitsgruppe "Entzündliche Erkrankungen des Zentralnervensystems" (Frank Weber) und einer Forschungsgruppe der Universität Dresden (Tjalf Ziemssen) zusammen. Hierbei stellte sich heraus, dass - unabhängig von der Art der experimentellen Induzierung der Krankheit im Tiermodell - ein konstanter Satz von Proteinmarkern existiert, mit dem krankes von gesundem Gewebe zuverlässig unterschieden werden kann. Die Wissenschaftler wollen nun den diagnostischen Wert dieser Marker in menschlichen Gehirnproben von Verstorbenen und im Liquor von Multiple-Sklerose-Patienten prüfen.
Ausblick
Die geschilderten proteomanalytischen Ansätze repräsentieren nur einen Teil der aktuellen experimentellen Strategien, die gegenwärtig am Max-Planck-Institut für Psychiatrie verfolgt werden. Nachdem die Sequenzierung des menschlichen Genoms abgeschlossen ist, sind globale Analysen von Sequenzvariationen in menschlichen Populationen möglich geworden. Diese Variationen sind nicht nur für die individuellen Unterschiede zwischen den Menschen verantwortlich, sondern bestimmen auch die Anfälligkeit für Krankheiten. Die Mehrheit dieser Variationen beruht auf so genannten SNPs (Single Nucleotide Polymorphisms), bei denen einzelne Basenpaare in der DNA-Sequenz verändert sind. Um diese Unterschiede näher zu charakterisieren, entstand am Max-Planck-Institut für Psychiatrie in Zusammenarbeit mit der Firma GlaxoSmithKline das Genetics Research Centre. Dort können SNP-Analysen mithilfe von Hochdurchsatz-Verfahren durchgeführt werden.
Die Analyse der mitochondrialen RNA-Expression wurde ebenfalls am Institut etabliert. Diese Methode erfasst quantitativ die von den Genen transkribierten RNA-Moleküle (Abb. 1), wobei für affektive Störungen insbesondere die Genexpression und damit die Genaktivität in Gehirnarealen interessant ist. Im Tiermodell konnten Mitarbeiter bereits wichtige Marker-RNAs identifizieren, die eine Prädisposition für Angstzustände anzeigen.
Die Gesamt-Analyse der Proteine, die Proteomik, reiht sich in diese empirisch orientierten Ansätze ein. Ein wichtiges Ziel der Proteomikstudien am Institut für Psychiatrie ist die zuverlässigere Diagnose von Gehirnkrankheiten, für die bislang noch keine molekularen Marker in der Klinik zur Verfügung stehen. Derartige Untersuchungen sind die Voraussetzung für Datenbanken über Krankheitsmechanismen auf der Ebene der Proteine, die wiederum erst die Entwicklung von individualspezifischen Therapien ermöglichen. Hinzu kommt, dass fast alle Medikamente, die derzeit auf dem Markt sind, auf Proteine als Zielmoleküle einwirken. Die vergleichsweise geringe Zahl von Zielmolekülen, die der Pharmakotherapie bisher zur Verfügung stehen, wird durch die beschriebenen Hochdurchsatz-Verfahren in den nächsten Jahren um ein Vielfaches erweitert werden. Die Verbindung zwischen Humangenetik, chemischer Genomik und Proteomik ermöglicht die Entwicklung neuer pharmakologischer Wirkmechanismen und schafft so eine Zukunftsperspektive für die Prävention und Therapie von Erkrankungen des zentralen Nervensystems. Das Max-Planck-Institut für Psychiatrie will zu dieser Entwicklung im Bereich Depression, Angst und neurologischen Krankheiten einen wichtigen Beitrag leisten.


