Forschungsbericht 2013 - Max-Planck-Institut für molekulare Genetik
Gibt es zwei Klassen von Promotoren?
Are there two classes of promoters?
Einleitung
Nachdem über viele Jahre das Interesse der Genomforschung auf der Bestimmung der Gene gelegen hat, rückte im letzten Jahrzehnt die Frage in den Mittelpunkt, wie welches Gen zu einem bestimmten Zeitpunkt oder unter bestimmten Bedingungen an- oder abgeschaltet wird. Diese Kontrollebene wird als Genregulation bezeichnet. Sie ist verantwortlich dafür, dass in den verschiedenen Zellen eines Organismus unterschiedliche Proteine erzeugt werden oder dass eine bestimmte Zelle zu unterschiedlichen Zeiten unterschiedliche Proteine erzeugt, um zum Beispiel auf eine Infektion mit einer Immunantwort zu reagieren.
Die Information, wann welches Gen exprimiert, das heißt, in ein Protein übersetzt werden soll, ist bis zu einem gewissen Grad in der Buchstabenfolge des Genoms kodiert – allerdings nicht in der systematischen Art und Weise, in der die Proteine durch den genetischen Code definiert werden. Das An- oder Abschalten von Genen geschieht durch sogenannte Transkriptionsfaktoren. Dies sind ebenfalls Proteine, die an die DNA unter anderem vor dem zu regulierendem Gen andocken und dessen Expression aktivieren oder unterdrücken können. In dem Forschungsbericht "Untersuchung von Bindungsstellen zur Aktivierung von Genen" für das Jahrbuch der Max-Planck-Gesellschaft 2005 wurde die Funktionsweise der Transkriptionsfaktoren beschrieben [1]. Diejenigen Bereiche im Genom, in denen sich die Bindungsstellen der Transkriptionsfaktoren hauptsächlich befinden, werden als Promotoren und Enhancer bezeichnet. Promotoren liegen nahe am Beginn eines Gens und kontrollieren es genau. Enhancer sind in ihrer Position zum regulierten Gen frei und können unter Umständen bis zu einer Million Basenpaaren entfernt liegen. Häufig befinden sich sogar noch andere Gene zwischen einem Enhancer und dem von ihm kontrollierten Gen.
Der Beitrag im diesjährigen Jahrbuch konzentriert sich auf die Promotoren im menschlichen Genom. In der Literatur werden im Allgemeinen zwei mögliche Charakteristika der Promotoren betont. Zum einen gibt es das TATA-binding protein (TBP), das an viele dieser Promotoren bindet. Sie verfügen alle über einen bestimmten Sequenzabschnitt, die TATA-Box – eine Abfolge der Basenpaare TATA -, an die TBP bindet. Ein anderes Charakteristikum vieler humaner Promotoren ist die sogenannte CpG-Insel (CpG island, CGI). Sie bezeichnet einen Sequenzbereich, in dem die Basenpaarfolge CG signifikant häufiger vorkommt als in anderen Bereichen der DNA. Das humane Genom nutzt dieses Dinukleotid eigentlich selten, sodass eine Häufung von CG-Abfolgen statistisch hervorsticht. Das „p“ in CpG betont die Phosphatbindung zwischen den Basen, um eine Verwechslung mit der komplementären Abfolge GC auszuschließen.
Promotoren unterscheiden sich im CpG-Gehalt
Abbildung 1 zeigt ein Histogramm mit der Dichte an CpGs in den Sequenzen der menschlichen Promotoren. Man würde erwarten, dass die Dichte an CpGs um einen Mittelwert gleichmäßig variiert. Statt dessen sieht man zwei Klassen von Promotoren, nämlich solche mit einer hohen Dichte an CpGs und solche mit niedrigem Gehalt an CpGs [2]. Diese Beobachtung wirft eine Reihe von Fragen auf, vor allem die nach den regulatorischen Mechanismen in den beiden Klassen von Promotoren. Funktionieren die beiden Promotorklassen nach dem gleichen Muster oder gibt es unterschiedliche regulatorische Mechanismen?
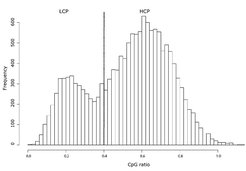
Es gibt eine Reihe weiterer Eigenschaften von Genen und Promotoren, die mit der gezeigten Zweiteilung der Promotoren einhergehen. Zuerst ist festzustellen, dass die High-CpG-Promotoren (HCPs) genau jene Promotoren sind, die über eine CpG-Insel verfügen. Umgekehrt haben viele der Low-CpG-Promotoren (LCPs) eine TATA-Box-Bindungsstelle. Die TATA-Box gilt allgemein als ein Kennzeichen von gewebespezifisch exprimierten Genen. Abbildung 2 zeigt das Auftreten einer TATA-Box in den Promotoren von Genen mit LCP-Promotoren, die in verschiedenen Geweben exprimiert werden [3]. Die Höhe der Balken symbolisiert die Häufigkeit der vorhergesagten TATA-Box. Die rechte ebene Dimension zeigt die Sequenz in der Umgebung des Startpunkts der Transkription, also den Übergang vom Promoter in den kodierenden Bereich des betreffenden Gens. Negative Zahlen beziehen sich auf den DNA-Bereich vor dem Gen, TSS bezeichnet den Transkriptionsstart und positive Zahlen bezeichnen Positionen in bereits kodierenden Regionen des Gens. Auf der linken Achse sind diejenigen Gewebe benannt, für die Gene beziehungsweise Promotoren ausgewählt und analysiert wurden. Zum Beispiel sieht man bei Genen, die in der Lunge exprimiert werden, unmittelbar vor dem Transkriptionsstart einen hohen gelben Balken, also eine klare Präferenz für eine TATA-Box an dieser Stelle in diesen Genen. Die Abbildung insgesamt illustriert die Bedeutung der TATA-Box bei der Regulation von gewebespezifisch exprimierten LCP Genen. Die gleiche Analyse für HCP-Gene, auch wenn diese gewebespezifisch exprimiert sind, zeigt ein weitaus schwächeres Signal.
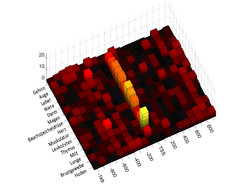
Gewebespezifische Regulation
Neben der TATA-Box mit ihrem Bindungsprotein TBP gibt es zahlreiche weitere sequenzspezifisch bindende Transkriptionsfaktoren. Als ein Beispiel sei hier MEF2 erwähnt, der eine wichtige Rolle bei der Regulation der Gene in Muskulatur und im Herzmuskelgewebe spielt. Abbildung 3 zeigt in gleicher Weise wie Abbildung 2 die Prominenz der MEF2-Bindungsstellen in LCP-Genen verschiedener Gewebe. Man sieht, dass nur diejenigen Gene, die nachweislich in Muskulatur oder Herzmuskelgewebe exprimiert werden, die MEF2-Bindungsstelle enthalten. Wird die entsprechende Analyse bei HCP-Genen durchgeführt, fehlt grundsätzlich diese Bindungsstelle und es gibt dementsprechend keinen Unterschied zwischen ihrem Auftreten in denjenigen Genen, die in verschiedenen Geweben exprimiert sind.

Epigenetische Regulation
Die beschriebenen Beobachtungen deuten auf unterschiedliche Kontrollmechanismen der LCPs und HCPs hin. Unterstützt wird diese Annahme durch einen Blick auf eine weitere Ebene der Genregulation. Neben den Sequenzmustern, die von Transkriptionsfaktoren erkannt werden, gibt es biochemische Markierungen an den sogenannten Histonproteinen, die über weite Strecken die DNA bedecken. Diese Markierungen können Acetylierungen, Methylierungen oder andere Modifikationen sein. Summarisch werden sie als Histonmodifikationen bezeichnet, ihre genaue Beschreibung ergibt sich aus dem betreffenden Histon und der Position und Art der Modifikation (zum Beispiel Histon3-Lysin4-Acetylierung, H3K4ac). Man hat festgestellt, dass die Markierungen mit dem Aktivitätsstatus eines Promotors einhergehen. Daraus folgt, dass aus den Markierungen, die sich in einem Promotor befinden, über mathematische Methoden die Expression eines Gens vorhergesagt werden kann.
Es gibt eine Vielzahl solcher Modifikationen, von denen über 30 entlang des Genoms biochemisch in ihrer Stärke gemessen wurden. Mithilfe eines mathematischen Modells gelang es den Bioinformatikern des Max-Planck-Instituts für molekulare Genetik zu bestimmen, welche Modifikationen die meisten Informationen über die Expression der Gene tragen [4]. Wenn aber die Unterscheidung der Promotoren in HCPs und LCPs tatsächlich so fundamental ist, wie die Wissenschaftler meinen, sollte sich diese Zweiteilung auf der Ebene der Modifikationen wiederfinden. Um dies zu überprüfen, stellten sie das mathematische Modell separat für die beiden Gruppen von Promotoren auf und bestimmten jeweils die informationstragenden Histonmodifikationen. In der Tat konnten sie nachweisen, dass unterschiedliche Gruppen von Histonmodifikationen mit der Aktivierung in HCPs und LCPs verbunden sind. Dies erhärtet die Annahme, dass die beiden Gruppen von Promotoren unterschiedlich reguliert werden.
Noch sind die regulatorischen Mechanismen zur Expression von Genen nicht vollständig verstanden. Die Forscher der Abteilung Bioinformatik haben aber Hypothesen aufgestellt, wie sie funktionieren könnten. Von den HCPs ist bekannt, dass die RNA-Polymerase, also jenes Enzym, das für die Transkription zuständig ist, die meiste Zeit am Promoter der HCP-Gene präsent ist - unabhängig davon, ob das Gen transkribiert wird oder nicht. Bei den LCPs verhält sich die RNA-Polymerase anders. Wenn ein von einem LCP kontrolliertes Gen nicht aktiv ist, befindet sich auch keine RNA-Polymerase an dessen Promoter. Dieser (weitere) Unterschied lässt vermuten, dass die Kommandos, die zur Aktivierung eines HCP-Gens führen, nur den eigentlichen Start des Transkriptionsvorgangs einleiten. Bei einem LCP-Gen hingegen muss vor der Aktivierung zuerst die Transkriptionsmaschinerie an ihren Einsatzort, den LCP-Promotor, geholt werden. Dies könnte der Hintergrund für die unterschiedlichen Histonmodifikationen sein. Gleichzeitig nimmt man an, dass HCP-Gene häufig in vielen Geweben exprimiert werden - folglich ist bei ihnen die gewebespezifische Regulation weniger wichtig als bei LCPs.
Die heute verfügbaren Sequenzdaten zusammen mit einer Vielzahl biochemischer Messungen ermöglichen Analysen wie die hier beschriebenen. In der Summe lassen sich daraus Hypothesen über biochemische Mechanismen aufstellen oder untermauern, die dann wiederum zu neuen Experimenten und weiteren Einsichten führen. Diese Form des Studiums der Genregulation wird regulatory genomics genannt und ist eines der aktuellen und spannendsten Gebiete der Molekulargenetik und Genomforschung.


