Forschungsbericht 2009 - Max-Planck-Institut für terrestrische Mikrobiologie
Etablierung der kompatiblen Interaktion von Ustilago maydis mit seiner Wirtspflanze Mais
Organismische Interaktionen (Kahmann) (Prof. Dr. Regine Kahmann)
MPI für terrestrische Mikrobiologie, Marburg
Einleitung
Die Brandpilze (Ustilaginomycotina) sind eine mehr als 1200 Arten umfassende Gruppe von pflanzenpathogenen Pilzen, die zahlreiche Getreidearten befällt, darunter Weizen, Mais, Gerste und Hafer. Der Brandpilz Ustilago maydis parasitiert auf Mais (Zea mays) sowie dessen Urform Teosinthe (Zea mays ssp. parviglumis) und ist der Verursacher des Maisbeulenbrandes. Dank seiner einfachen Kultivierbarkeit, des kurzen Infektionszyklus von zirka zwei Wochen sowie der hervorragenden Zugänglichkeit für molekulargenetische Techniken, wurde U. maydis in den letzten Jahren als Modellsystem sowohl für zellbiologische als auch pathogenitäts-relevante Fragestellungen etabliert. Seit 2006 ist auch die komplette Genomsequenz von U. maydis bekannt und öffentlich verfügbar [1].
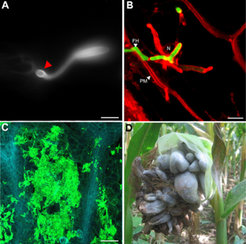
Die Infektion der Wirtspflanze Mais beginnt mit der direkten Penetration der Epidermis. Dabei werden spezielle Infektionsstrukturen, die so genannten Appressorien, ausgebildet (Abb. 1A). Die penetrierende Hyphe dringt in die Wirtszelle ein, wo sie allerdings von der unverletzt bleibenden Plasmamembran der Pflanzenzelle umhüllt bleibt (Abb. 1B). In dieser biotrophen Interaktion bleiben die infizierten Pflanzenzellen während des gesamten Infektionszyklus am leben und ernähren die wachsenden Pilzzellen. Im weiteren Verlauf der Infektion kommt es zur massiven Proliferation der Pilzhyphen in den Zellzwischenräumen (Abb. 1C; [2]) und schließlich zur Ausbildung von Pflanzentumoren (Abb. 1D), in denen der Pilz seinen Lebenszyklus durch die Bildung der so genannten Teliosporen abschließt [3].
Unterdrückung der pflanzlichen Abwehr durch U. maydis
Frühe Symptome einer U. maydis Infektion sind Chlorosen der Blattoberfläche und, in seltenen Fällen, kleine nekrotische Bereiche am Ort der Infektion. In dieser Phase der Interaktion weisen U. maydis – Hyphen polares Spitzenwachstum auf und bewegen ihr gesamtes Cytoplasma zur Hyphenspitze. Ältere Hyphenabschnitte erscheinen dabei leer und werden durch Septen abgetrennt [4]. Pflanzenzellen, die solche kollabierten Hyphen enthalten, reagieren oft mit programmiertem Zelltod als Abwehrmaßnahme gegen den Pilz. In einer späteren Infektionsphase induziert U. maydis die Bildung von Tumoren durch Vergrößerung und Proliferation der Pflanzenzellen. In diesen Tumoren aggregieren pilzliche Zellen, ohne Zelltod im umgebenden pflanzlichen Gewebe auszulösen [2, 5].
Untersuchungen der transkriptionellen Antwort von Mais auf eine Infektion durch U. maydis mit dem Affymetrix© Microarray-System zeigten eine frühzeitige Erkennung des Pilzes auf der Blattoberfläche, die eine starke, unspezifische Pflanzenabwehrreaktion zur Folge hat. Mit Etablierung der biotrophen Interaktion zirka 24 Stunden nach der Infektion nimmt diese Abwehrreaktion wieder ab [5]. Gleichzeitig werden in der Pflanze Gene für die Zelltod-Unterdrückung induziert, wie beispielsweise der Bax-Inhibitor-1 [6]. Diese Befunde legen nahe, dass U. maydis nach erfolgreicher Etablierung der biotrophen Interaktion die Pflanzenabwehr, insbesondere den programmierten Zelltod, aktiv unterdrücken kann und damit die Ausbildung einer kompatiblen Interaktion mit der Wirtspflanze ermöglicht.
Das Effektorprotein Pep1
Um den pflanzlichen Abwehrreaktionen entgegenzuwirken, sekretieren mikrobielle Pathogene eine Vielzahl von Proteinen, so genannte Effektoren, welche in die Pflanzenabwehr eingreifen und so die erfolgreiche Infektion ermöglichen [7]. Mikrobielle Effektorproteine weisen eine hohe strukturelle und funktionelle Vielfalt auf. Oftmals handelt es sich um Proteine ohne konservierte funktionelle Domänen, die meist keinerlei Ähnlichkeit zu bereits bekannten Proteinen haben.
Das Genom von U. maydis kodiert 386 putativ sekretierte Proteine, denen gemäß ihrer Aminosäuresequenz keine enzymatische Funktion zugeschrieben werden kann. Interessanterweise sind viele der Gene, die solche potenzielle Effektoren kodieren, in Genclustern arrangiert – sie liegen also in räumlicher Nähe zueinander [1]. U. maydis Mutanten, in denen diese Gencluster gezielt ausgeschaltet wurden, zeigten verschiedene Defekte während der pathogenen Entwicklung, waren jedoch sämtlich zur erfolgreichen Besiedelung der penetrierten Wirtszelle fähig [1]. Dies führte zu der Vermutung, dass es weitere Effektoren außerhalb der beschriebenen Gencluster geben müsse, die initial die Etablierung der kompatiblen Interaktion und somit auch die beobachtete Suppression der pflanzlichen Abwehrreaktion vermitteln.
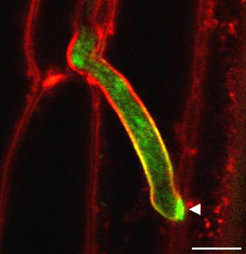
Eine systematische Analyse von mutmaßlichen Effektorgenen in U. maydis führte schließlich zur Identifikation des neuartigen Effektorproteins Pep1 (protein essential during penetration 1). Deletionsmutanten von Pep1 weisen einen völligen Verlust der Tumorbildung in infizierten Maispflanzen auf. Auch für einen weiteren Brandpilz, den Erreger des Gerstenhartbrandes (Ustilago hordei) ist Pep1 ein essenzieller Pathogenitätsfaktor [6]. Mittels konfokaler Mikroskopie konnte gezeigt werden, dass Pep1-Deletionsmutanten im Gegensatz zum Wildtyp 24 Stunden nach Infektion die pflanzliche Plasmamembran invaginieren, jedoch nicht zur weiteren Besiedelung des Wirtsgewebes in der Lage sind [8]. Um das Pep1-Protein im Pflanzengewebe zu lokalisieren, wurden fluoreszenzmarkierte Versionen des Proteins genutzt. GFP- (green fluorescent protein) markiertes Pep1 wird demnach von intrazellulär wachsenden U. maydis Zellen sekretiert und akkumuliert an den Hyphenspitzen, vor allem während der Zell-Zell-Passagen (Abb. 2). Zudem konnte gezeigt werden, dass sich Pep1 an der Schnittstelle zwischen Pilzhyphe und Wirtszelle, der so genannten biotrophen Interaktionszone, befindet [8].
Der Verlust der Pathogenität der Pep1-Mutante geht einher mit makroskopischen Symptomen: dem Absterben infizierter Blattbereiche, einer erhöhten Autofluoreszenz der Zellwand sowie der Akkumulation von Wasserstoffperoxid um die Appressorien herum [8]. Microarray- Analysen von Maispflanzen 24 Stunden nach Infektion mit Δpep1-Mutanten zeigten zudem, dass es im Gegensatz zur Interaktion mit dem Wildtyp nicht zu einer Reprimierung der Pflanzenabwehr kommt. Die Antwort der Pflanze auf eine Infektion durch Δpep1-Mutanten ist somit typisch für eine Nicht-Wirts-Antwort, also für eine Interaktion mit einem Pathogen, das nicht auf eine erfolgreiche Besiedelung von Mais adaptiert ist. Oder mit anderen Worten: Die Sekretion von Pep1 in die biotrophe Interaktionszone ist eine essenzielle Voraussetzung für die Etablierung einer erfolgreichen Interaktion zwischen Ustilago maydis und seiner Wirtspflanze, dem Mais. Bisher unbekannt ist allerdings, wie Pep1 in der Pflanze funktioniert. Denkbar wäre beispielsweise, dass Pep1 grundlegende Komponenten der pflanzlichen Abwehr inhibiert, aber auch weitere Erklärungsmodelle erscheinen plausibel (Abb. 3).
Da es sich bei Pep1 offenbar um einen grundlegenden Faktor für die Pathogenität von Brandpilzen handelt, ist es von größtem Interesse, die Funktionsweise von Pep1 auf molekularer Ebene zu verstehen. Das umfassende Verständnis der molekularen Mechanismen, die der Interaktion von pathogenen Pilzen mit ihren Wirtspflanzen zu Grunde liegen, ist dabei nicht allein von wissenschaftlicher Relevanz. In Anbetracht der weltweiten, jährlich wiederkehrenden Ernteausfälle durch Schadpilze und des stetig steigenden Einsatzes von Umwelt und den Menschen gefährdenden Fungiziden besteht die dringende Aufgabe, dieses Wissen für die Züchtung stabiler Resistenzen gegen Schadpilze in Getreidepflanzen zu nutzen.



