Forschungsbericht 2006 - Max-Planck-Institut für Dynamik komplexer technischer Systeme
Die Kontrolle der Zelldifferenzierung: Neue Methoden zur Erforschung der Struktur und Funktion eines Molekularen Netzwerkes
Fachgruppe: Moleculare Netzwerkanalyse (Prof. Dr. Wolfgang Marwan)
MPI Dynamik komplexer technischer Systeme, Magdeburg
Mit den bahnbrechenden Erfolgen der Genomforschung stellt sich die Frage nach der funktionellen Wechselwirkung der zahlreichen Genprodukte bei der Kontrolle komplexer zellulärer Funktionen. Die Steuerung der Entwicklung einer Stammzelle zur terminal ausdifferenzierten Zelle, zum Beispiel, erfolgt durch ein Wechselspiel von Proteinen und Nucleinsäuren, Molekülen, die im Verbund eines komplexen molekularen Netzwerkes wirken. Ein molekulares Netzwerk lässt sich sowohl als System gekoppelter, zum Teil verzweigter biochemischer Reaktionen, als auch als mathematische Struktur im Sinne eines Hypergraphen begreifen. Der Wechsel zwischen beiden Betrachtungsweisen kann helfen, den Aufbau eines regulatorischen Netzwerkes aufzuklären und die regulatorische Funktion der wechselwirkenden Moleküle zu ermitteln. Die gezielte, mathematisch berechenbare Beeinflussung bestimmter Teile eines Netzwerkes könnte ein weitergehendes Ziel sein, denn viele Krankheiten stehen mit einer Fehlregulation der Genexpression und/oder der Verarbeitung zellulärer Signale in ursächlichem Zusammenhang.
Die grundlagenorientierten Arbeiten zur Analyse molekularer Netzwerke fokussieren sich auf zwei Fragen:
• Wie lässt sich die regulatorische Architektur und die funktionelle Dynamik eines Netzwerkes durch systematische Untersuchung seiner Komponenten und ihrer funktionellen Wechselwirkung ermitteln und an der lebenden Zelle quantitativ und zeitaufgelöst erfassen?
• Wie lässt sich die funktionelle Struktur eines Netzwerkes auf der Grundlage experimenteller Daten mathematisch eindeutig beweisen?
Im vorliegenden Beitrag werden experimentelle und theoretische Arbeiten zu diesem Problemkreis am Beispiel eines eukaryontischen Modellsystems, der Zelldifferenzierung (Sporulation, d.h. Fruchtkörperbildung) von Physarum polycephalum, vorgestellt. Es wird gegenwärtig eine „Gene Discovery Pipeline“ zur systematischen Identifizierung von Genen etabliert, die an der Kontrolle der Sporulation beteiligt sind und es werden ihre regulatorischen Wechselwirkungen mithilfe einer systematisch anwendbaren Methode erforscht [2].
Die Sporulation von Physarum polycephalum, Modellsystem eines sensorisch kontrollierten Differenzierungsprozesses
Obwohl alle Zellen eines vielzelligen Organismus, gleich ob Tier oder Pflanze, das identische genetische Material besitzen, unterscheiden sich die Zellen als elementare Bausteine der verschiedenen Gewebe und Organe dennoch hinsichtlich Form, Funktion und biochemischer Zusammensetzung. Diese Unterschiede erklären sich aus dem jeweiligen Satz von Genen, der zelltypspezifisch abgelesen (exprimiert) wird. Die Expression dieser Gene, die damit verbundenen Differenzierungsprozesse, und das Verhalten der Zelle hinsichtlich Überleben, Zellteilung oder Apoptose (Zelltod) wird durch exogene Signale aus der unmittelbaren oder der weiteren Umgebung des Zellverbandes gesteuert. Exogene Signale dienen der Zell-Zell-Kommunikation, werden auf dem Hintergrund der Entwicklungsgeschichte einer jedweden Zelle interpretierend verarbeitet und steuern so das Verhalten der Zellen, aus denen ein Organismus besteht. Dies gilt für die Embryonalentwicklung ebenso, wie für die Homöostase der Gewebe und Organe oder für die Regeneration, wie sie zum Beispiel bei der Wundheilung besonders eindrucksvoll in Erscheinung tritt.
Der als Modellsystem dienende Eukaryont Physarum polycephalum differenziert während seines Entwicklungscyclus zu sieben unterschiedlichen Zelltypen. In einem dieser Entwicklungsstadien entsteht eine vielkernige Riesenzelle, ein so genanntes Plasmodium. Das Plasmodium hat einerseits die Fähigkeit zu unbegrenztem Wachstum und kann andererseits unter bestimmten Bedingungen zwei alternative Entwicklungswege beschreiten (Sporulation oder Spherulation, d.h. Bildung von Dauerstadien). Durch kombinierte Einwirkung von Licht und Nahrungsmangel verliert die Riesenzelle ihr unbegrenztes replikatives Potenzial und differenziert unter Ausbildung einer Vielzahl von Fruchtkörpern (Sporulation). Unter Laborbedingungen kann der Differenzierungsprozess durch einen Puls von dunkelrotem Licht ausgelöst werden.
Nicht nur die Sporulation, sondern der gesamte Entwicklungscyclus von Physarum ist experimentell gut kontrollierbar (Abb. 1), wodurch wir das Methodenrepertoire der klassischen und molekularen Genetik einsetzen können, um die molekularen Komponenten zu identifizieren, welche die Differenzierung der Zelle steuern.
![Entwicklungscyclus des Eukaryonten Physarum polycephalum und seine Anwendung für die Genetik und Molekularbiologie. Die aus Sporen (Spo) schlüpfenden haploiden (mit einfachem Chromosonensatz) Amöben (Am) unterschiedlicher Paarungstypen verschmelzen zu einer diploiden(mit doppeltem Chromosonensatz) Zygote (Zy), die sich zu einem vielkernigen, diploiden Plasmodium entwickelt [1], das schließlich nach Belichtung sporulieren kann. In den Fruchtkörpern (Spgm) kommt es nach einer Meiose (Reifeteilung) zur Ausbildung haploider Sporen, wodurch sich der Entwicklungscyclus schließt. Mithilfe einer temperatursensitiven Mutante kann man aus einer haploiden Amöbe unmittelbar ein haploides Plasmodium (hPl) züchten. Auf diese Weise ist es möglich, Mutanten im haploiden Hintergrund zu erzeugen (innerer Cyclus) und haploide Plasmodien ohne vorherige Rückkreuzung phänotypisch zu screenen. Die erhaltenen Mutanten können dennoch genetisch analysiert werden, da die ungeschlechtliche Entwicklung haploider Plasmodien durch Erhöhung der Temperatur auf 30 °C experimentell blockiert werden kann, und die Mutanten dann mit einem anderen Amöbenstamm gekreuzt werden können. Nach einer Meiose der Kerne des diploiden Plasmodiums kann man aufgrund der Cosegregation molekularer Marker mit dem Phänotyp der Mutante das betroffene Gen kartieren und molekular identifizieren.](/444480/original-1293749353.jpg?t=eyJ3aWR0aCI6MjQ2LCJvYmpfaWQiOjQ0NDQ4MH0%3D--c4a5b434fc79bc4eec4d21b68b527d97a35b9ca1)
Das Netzwerk im Baukasten-System: Zeitaufgelöste somatische Komplementationsanalyse
Wenn sich zwei Plasmodien berühren, verschmelzen sie von selbst, wobei sich ihr Cytoplasma mit den darin enthaltenen Zellkernen vermischt. Mischen sich die Cytoplasmata zweier Mutanten, die Defekte (Blockaden) an unterschiedlichen Stellen des Sporulations-Kontroll-Netzwerkes tragen, so kann die Sporulation dennoch in normaler Weise ablaufen. Die so genannte Komplementation beider Gendefekte beruht darauf, dass ein Partner das funktionsfähige, im jeweils anderen Partner defekte Genprodukt beisteuert und umgekehrt: Die beiden, an verschiedenen Stellen defekten Netzwerke ergänzen sich zu einem intakten, funktionsfähigen Netzwerk, das Signale aufnehmen, korrekt verarbeiten und so die Differenzierung der Zelle steuern kann [3].
Die genetische Komplementation von Gendefekten durch Kreuzung zweier Individuen ist ein klassisches Phänomen der Genetik. Die somatische Komplementation von Gendefekten durch Zellfusion ermöglicht darüber hinaus eine neuartige und weitergehende Analyse eines Netzwerkes, weil der Zeitpunkt, zu dem die Komplementation durch Mischen der Genprodukte erfolgt, beliebig variiert werden kann (Abb. 2a und 2b).
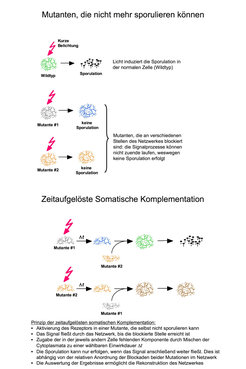
Dadurch ist es möglich, den Weg, den ein in das Netzwerk eingespeistes Signal nimmt, Schritt für Schritt nachzuvollziehen. Die chemische Mutagenese, mit deren Hilfe man praktisch jede der beteiligten Komponenten mit vergleichbarer Wahrscheinlichkeit ausschalten kann, ermöglicht im Konzept die fast lückenlose Bestimmung aller Knoten aus denen das Netzwerk geknüpft ist. Die Komplementation von Gendefekten durch Zellfusion erlaubt mithilfe rechnerischer Verfahren Rückschlüsse sowohl auf die Struktur des Netzwerkes als auch auf die Kinetik der Signalverarbeitung [3, 6]. Ausgangspunkt für die Entwicklung dieser Verfahren war die Entdeckung, dass sich die signalverarbeitende Aktivität eines Proteins und damit sein reaktionskinetisches Verhalten in einer lebenden Zelle analysieren lässt, ohne dass die komplexen regulatorischen Wechselwirkungen der anderen molekularen Komponenten des Netzwerkes bekannt sein müssen [4, 5]. Dies ermöglicht eine schrittweise Erforschung des Netzwerkes, ohne dass die lückenlose Kenntnis aller Komponenten a priori erforderlich wäre.
Die Rekonstruktion des Netzwerkes erfolgt mit Hilfe eines Petri-Netzes [6] (Abb. 3).
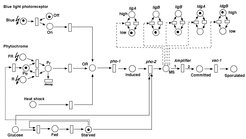
Ein Petri-Netz ist eine mathematisch definierte Struktur, die sich graphisch anschaulich darstellen lässt, aber auch in Tabellenform von einem Computer erzeugt, gelesen und ausgewertet werden kann. Die Regeln, die bei der Erstellung eines Petri-Netzes gelten, ermöglichen die korrekte und folgerichtige Darstellung jeder denkbaren biochemischen Reaktion oder genetischen Wechselwirkung und erlauben zudem die Wiedergabe hierarchisch strukturierter kausaler Verknüpfungen.
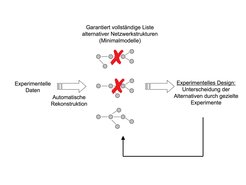
Die automatische Netzwerk-Rekonstruktion: Strukturbeweis durch Ausschluss
Petri-Netze sind auch hilfreich, um Netzwerke auf der Grundlage experimenteller Daten automatisch zu rekonstruieren. Bei der herkömmlichen, in den Biowissenschaften üblichen Vorgehensweise, interpretiert man experimentelle Ergebnisse, um zu einer plausiblen, widerspruchsfreien Modellvorstellung zu kommen, die möglichst mit allen experimentellen Befunden im Einklang steht. Insbesondere bei der Erforschung komplexer Prozesse ist aber schwer auszuschließen, dass alternative Erklärungsmöglichkeiten existieren, die die experimentellen Befunde ebenso gut oder vielleicht sogar besser wiedergeben könnten als dies das favorisierte Modell tut. Es ist eine Lehrbuch-Weisheit der Physikalischen Chemie, dass ein kinetisches Modell einen (bio-) chemischen Mechanismus bestenfalls widerlegen, nie aber beweisen kann. Die automatische Netzwerk-Rekonstruktion bietet hier einen Fortschritt: Allein auf der Grundlage von experimentellen Daten und unabhängig von menschlicher Intuition oder Vorstellungskraft, berechnet der Computer eine garantiert vollständige Liste aller alternativen minimalen Netzwerke, die den Datensatz zu erklären vermögen. Das Verfahren wurde in enger Kooperation mit Professor Robert Weismantel und Dr. Annegret Wagler vom Institut für Mathematische Optimierung der Universität Magdeburg entwickelt. Mithilfe eines geeigneten experimentellen Datensatzes erhält man eine vom Computer erzeugte Liste von wenigen alternativen minimalen Netzwerk-Strukturen, die durch wenige, gezielte Experimente unterscheidbar sind. Aufgrund der durch mathematischen Beweis garantierten Vollständigkeit der Liste resultiert ein Strukturbeweis durch Ausschluss der nichtzutreffenden alternativen Erklärungsmöglichkeiten (Abb. 4).
![Entwicklungscyclus des Eukaryonten Physarum polycephalum und seine Anwendung für die Genetik und Molekularbiologie. Die aus Sporen (Spo) schlüpfenden haploiden (mit einfachem Chromosonensatz) Amöben (Am) unterschiedlicher Paarungstypen verschmelzen zu einer diploiden(mit doppeltem Chromosonensatz) Zygote (Zy), die sich zu einem vielkernigen, diploiden Plasmodium entwickelt [1], das schließlich nach Belichtung sporulieren kann. In den Fruchtkörpern (Spgm) kommt es nach einer Meiose (Reifeteilung) zur Ausbildung haploider Sporen, wodurch sich der Entwicklungscyclus schließt. Mithilfe einer temperatursensitiven Mutante kann man aus einer haploiden Amöbe unmittelbar ein haploides Plasmodium (hPl) züchten. Auf diese Weise ist es möglich, Mutanten im haploiden Hintergrund zu erzeugen (innerer Cyclus) und haploide Plasmodien ohne vorherige Rückkreuzung phänotypisch zu screenen. Die erhaltenen Mutanten können dennoch genetisch analysiert werden, da die ungeschlechtliche Entwicklung haploider Plasmodien durch Erhöhung der Temperatur auf 30 °C experimentell blockiert werden kann, und die Mutanten dann mit einem anderen Amöbenstamm gekreuzt werden können. Nach einer Meiose der Kerne des diploiden Plasmodiums kann man aufgrund der Cosegregation molekularer Marker mit dem Phänotyp der Mutante das betroffene Gen kartieren und molekular identifizieren. Entwicklungscyclus des Eukaryonten Physarum polycephalum und seine Anwendung für die Genetik und Molekularbiologie. Die aus Sporen (Spo) schlüpfenden haploiden (mit einfachem Chromosonensatz) Amöben (Am) unterschiedlicher Paarungstypen verschmelzen zu einer diploiden(mit doppeltem Chromosonensatz) Zygote (Zy), die sich zu einem vielkernigen, diploiden Plasmodium entwickelt [1], das schließlich nach Belichtung sporulieren kann. In den Fruchtkörpern (Spgm) kommt es nach einer Meiose (Reifeteilung) zur Ausbildung haploider Sporen, wodurch sich der Entwicklungscyclus schließt. Mithilfe einer temperatursensitiven Mutante kann man aus einer haploiden Amöbe unmittelbar ein haploides Plasmodium (hPl) züchten. Auf diese Weise ist es möglich, Mutanten im haploiden Hintergrund zu erzeugen (innerer Cyclus) und haploide Plasmodien ohne vorherige Rückkreuzung phänotypisch zu screenen. Die erhaltenen Mutanten können dennoch genetisch analysiert werden, da die ungeschlechtliche Entwicklung haploider Plasmodien durch Erhöhung der Temperatur auf 30 °C experimentell blockiert werden kann, und die Mutanten dann mit einem anderen Amöbenstamm gekreuzt werden können. Nach einer Meiose der Kerne des diploiden Plasmodiums kann man aufgrund der Cosegregation molekularer Marker mit dem Phänotyp der Mutante das betroffene Gen kartieren und molekular identifizieren.](/444480/original-1293749353.jpg?t=eyJ3aWR0aCI6MzQxLCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6NDQ0NDgwfQ%3D%3D--0d3379221477f228eb4173874420841448e6e8cc)


