Forschungsbericht 2005 - Max-Planck-Institut für Polymerforschung
Spinsondentechniken für weiche Nanostrukturen
Polymerspektroskopie (Prof. Dr. Hans Wolfgang Spiess)
MPI für Polymerforschung, Mainz
Abstandsmessungen im Nanometerbereich
Die Struktur eines Makromoleküls oder Molekülaggregats kann man dadurch aufklären, dass man hinreichend viele Abstände zwischen bekannten Punkten misst, ganz ähnlich wie man die Topographie eines Geländes durch Triangulation bestimmt. In einem Protein sind die bekannten Punkte bestimmte Aminosäurereste in der Sequenz, die durch gezielte Mutation adressiert werden können. In einem synthetischen Molekül bieten sich funktionelle Gruppen als adressierbare Fixpunkte an. Eine solche Strategie der Strukturaufklärung erfordert eine Technik, deren Messdaten ausschließlich von den Fixpunkten herrühren. Die Elektronenspinresonanz-Spektroskopie (ESR-Spektroskopie) erfüllt diese Forderung, indem sie Signale von ungepaarten Elektronen detektiert, die in Molekülen normalerweise nicht vorkommen, weil die chemische Bindung auf der Elektronenpaarbildung beruht. Allerdings ist es auch möglich, stabile freie Radikale mit einem ungepaarten Elektron zu erzeugen und sie gezielt als ESR-Sonden in makromolekulare Systeme einzubringen.
Die ungepaarten Elektronen der Sonden haben einen Spin, der mit einem kleinen magnetischen Moment verbunden ist. Wie ein Stabmagnet richten sie sich daher in einem äußeren Magnetfeld aus: vorzugsweise parallel zum Feld wie eine Kompassnadel, aber auch die antiparallele Ausrichtung hat nur eine geringfügig höhere Energie. Führt man diese Energiedifferenz durch Einstrahlung von Mikrowellen zu, so wird der Spin angeregt, der kleine Magnet klappt um und es kann ein Signal detektiert werden. Die erforderliche Mikrowellenfrequenz ist proportional zur Stärke des Magnetfeldes. Sie wird deshalb auch von der Anwesenheit weiterer Spins in der näheren Umgebung beeinflusst.
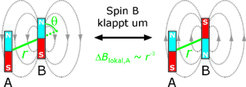
Ein zweiter Spin B erzeugt am beobachteten Spin A ein kleines Zusatzfeld. Dieses hängt von der Ausrichtung des Spins B, vom Winkel θ zwischen dem Spin-Spin-Vektor und dem Magnetfeld und nicht zuletzt vom Abstand r der beiden Spins ab (Abb. 1). Durch Weiterentwicklung des Puls-Doppelresonanzexperiments DEER [1] wurde die Genauigkeit der Messung des lokalen Zusatzfelds auf etwa 10 ppm des Gesamtfeldes verbessert. Dadurch können unter günstigen Bedingungen Abstände bis zu 8 nm, auf jeden Fall aber Abstände bis zu 5 nm gemessen werden [2]. So werden die typischen Dimensionen von Proteinen und synthetischen Makromolekülen zugänglich.
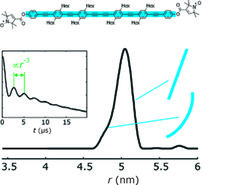
Auf derart kurzen Längenskalen ist weiche Materie inhärent flexibel. In Biomakromolekülen ist die Flexibilität bestimmter Molekülteile für die Funktion unabdingbar, zum Beispiel trifft das auf Schleifen in Proteinen zu. Deshalb erwartet man in der Regel eine Verteilung der Abstände zwischen Sondenpositionen, die durch den mittleren Abstand nur unzureichend charakterisiert würde. Um die vollständige Verteilung zu erhalten, wurden Analyseverfahren entwickelt, welche die Abhängigkeit vom Winkel θ aus den Daten eliminieren [3]. Dadurch wurde selbst die geringe Flexibilität gemeinhin als starr betrachteter Moleküle detektierbar, wie Abbildung 2 am Beispiel eines Phenylen-Ethynylen-Oligomers zeigt, das von Adelheid Godt von der Universität Bielefeld synthetisiert wurde. In der gleichen Kooperation wurden auch [2]Catenane untersucht, die jeweils aus zwei miteinander verschlungenen Ringen mit Durchmessern zwischen 3 und 7 nm bestehen. Die Analyse der Abstandsverteilung ergab hier, dass die Ringe in gewissen Lösungsmitteln expandiert und frei ineinander beweglich sind, während sie in anderer Umgebung Knäuel bilden.
Zwei Zustände eines Membranprotein-Komplexes
Die Messung von Abstandsverteilungen wird besonders interessant, wenn ein Molekül in mehreren verschiedenen Zuständen vorliegen kann. Das ist häufig für Proteine der Fall, deren Funktion auf einer Strukturänderung beruht, welche wiederum durch ein externes Signal induziert wird. Der pflanzliche Haupt-Lichtsammelkomplex IIb (LHCIIb), der in Kooperation mit Harald Paulsen von der Universität Mainz bearbeitet wird, absorbiert nicht nur Photonen und leitet die Anregungsenergie an photosynthetische Reaktionszentren weiter. Er ist auch in den Regelkreis involviert, der einen gleichen Durchsatz der Photosysteme I (Bereitstellung von Reduktionsäquivalenten) und II (Wasserspaltung) in Chloroplasten sicherstellt. Diese Regelung wird durch eine Phosphorylierung bzw. Dephosphorylierung des Threonin-Rests 5 erreicht. Im dephosphorylierten Zustand trimerisiert LHCIIb, hält sich vorzugsweise in Membranstapeln auf und versorgt Photosystem II mit Anregungsenergie. Nach einer Phosphorylierung dissoziieren die Trimere zu Monomeren und wandern in einzeln liegende Membranabschnitte, wo sie sich an Photosystem I anlagern. Die molekulare Basis dieser Präferenzänderung ist höchstwahrscheinlich eine Konformationsänderung der N-terminalen Schleife, in der sich Threonin 5 befindet. Zwar ist die Struktur von LHCIIb weitgehend aus Röntgenstrukturanalysen bekannt, allerdings ließ sich bisher nur das Trimer kristallisieren und selbst in den Kristallen sind die ersten 9-13 Reste der N-terminalen Schleife fehlgeordnet.
ESR-Messungen an gezielt spinmarkierten, detergenzsolubilisierten Monomeren liefern bimodale Abstandsverteilungen, wie man sie für ein Gleichgewicht zweier Konformationen erwartet.
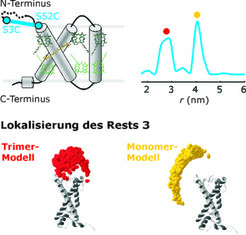
Durch eine Kombination mehrerer solcher Messungen und einer Messung am Trimer konnte die Position des Rests 3 in der N-terminalen Schleife trianguliert werden (Abb. 3) [4]. Dazu wurde bei der Modellierung die Kristallstruktur als Templat für den bekannten Teil der Struktur genutzt. Die Modelle legen eine Konformationsänderung nahe, bei der sich der N-Terminus von einer Position oberhalb des Proteinkerns im Trimer zu einer Position seitlich davon im Monomer bewegt. Interessanterweise ist eine ähnliche Änderung in Kristallstrukturen eines Annexins beobachtet worden, das durch Phosphorylierung seiner N-terminalen Schleife die Fähigkeit verliert, einzeln liegende Membranabschnitte zu Schichten zu aggregieren. Eine nähere Untersuchung in der Proteindatenbank hat gezeigt, dass die zugrunde liegenden Strukturmerkmale trotz der großen Variabilität der N-Termini von Annexinen über das Pflanzen- und Tierreich hinweg konserviert sind.
Radialverteilung von Gegenionen um einen Polyelektrolyten
Noch schwerer als flexible Strukturen im festen Aggregatzustand sind die hochdynamischen und schwach ausgeprägten Strukturen in Lösungen zu charakterisieren. In diesem Fall versagt auch die oben erläuterte Abstandsmessung zwischen Elektronenspins, da das Zusatzfeld durch die Reorientierung des Spin-Spin-Vektors auf Zeitskalen schneller als 10 μs zu Null ausgemittelt wird. Die schnelle Dynamik kann aber auch ausgenutzt werden, um einen Zugang zu Lösungsstrukturen zu erhalten. Dieser Zugang beruht auf dem Spin-Austausch zwischen kollidierenden Molekülen. Während der Kollision zweier stabiler Radikale überlappen deren Wellenfunktionen kurzzeitig stark und für diesen Zeitraum werden die beiden ungepaarten Elektronen ununterscheidbar. Trennen sich die Moleküle wieder, so erhalten sie deshalb nur mit 50-prozentiger Wahrscheinlichkeit ihr ursprüngliches Elektron zurück. Im anderen Fall ändert sich plötzlich die Umgebung des ungepaarten Elektrons und dadurch kommt es zu einer kleinen Verschiebung der Resonanzfrequenz. Im Mittel über die Zeit und alle Moleküle macht sich dieser Effekt als Linienverbreiterung bemerkbar. Das Ausmaß der Verbreiterung hängt von der Häufigkeit der Radikal-Radikal-Kollisionen und damit von der Radikalkonzentration ab.
Dieser Zusammenhang konnte nun ausgenutzt werden, um die Radialverteilung der Gegenionen um eine Polyelektrolytkette zu charakterisieren. Polyelektrolyte sind Polymere, in denen Ladungen einer Art auf den Wiederholeinheiten der Kette sitzen, während die entgegen gesetzten Ladungen von beweglichen Ionen getragen werden. In Lösung dissoziieren diese Ionen von der Kette. Es ergibt sich ein kompliziertes Wechselspiel von Kettenkonformation, elektrostatischer Abstoßung entlang der Kette und elektrostatischer Anziehung der Gegenionen, das zu einem interessanten Eigenschaftsspektrum von Polyelektrolytlösungen führt, aber derzeit theoretisch noch nicht vollständig beschrieben werden kann. So machen beispielsweise verschiedene Theorien unterschiedliche Aussagen zur Radialverteilung der Gegenionen, diese war aber bisher nicht direkt messbar.
Durch Einbringen eines kleinen Anteils spintragender Gegenionen konnte festgestellt werden, dass diese nur für sehr kurze Zeiträume unterhalb 1 ns Kontaktionenpaare mit den Wiederholeinheiten der Kette bilden, im Übrigen aber schnell diffundieren. In kettennahen Bereichen hoher lokaler Konzentration stoßen sie häufiger zusammen als in kettenfernen Bereichen. Die daraus resultierenden Linien verschiedener Breite überlagern sich zu einer charakteristischen Linienform, welche Information über die Radialverteilung enthält. Für einen flexiblen Polyelektrolyten in einem Wasser-Ethanol-Gemisch (Abb. 4) zeigte sich, dass die Linienform konsistent mit der Verteilung ist, die von einem Modell einer zylindrischen geladenen Zelle vorhergesagt wird.
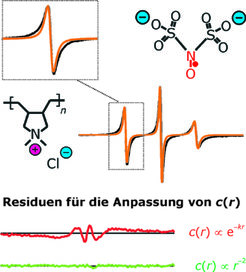
Die Voraussage eines exponentiellen Abfalls mit dem Abstand von der Kette durch ein einfacheres Modell ist dagegen im Widerspruch zur experimentellen Linienform. Die Übereinstimmung mit dem zylindrischen Modell legt nahe, dass die Kette infolge der Abstoßung ihrer gleichnamigen Ladungen zumindest auf einer Längenskala von bis zu 5 nm gestreckt ist [5].



