Forschungsbericht 2020 - Max-Planck-Institut für Infektionsbiologie
Mehr als ein Schaltplan: Wie wir die Grundlagen immunologischer Signalwege entschlüsseln wollen
Forschungsgruppe Visualisierung immunologischer Signalwege
Einführung
Unser Immunsystem ist eine Kommunikationsmaschine. Signale, die von Immunzellen empfangen und weitergegeben werden, sind entscheidend für die Unterscheidung zwischen Fremd und Selbst und damit für die Abwehr von Erregern. Dies gilt besonders für die angeborene Immunantwort, die innerhalb von Minuten auf Eindringlinge reagieren kann. Um unser Immunsystem zu verstehen, müssen wir diese Signalwege verstehen.
Komponenten ergeben noch keine Funktion
Die Entschlüsselung des menschlichen Genoms hat uns diesem Verständnis einen Schritt nähergebracht. In relativ kurzer Zeit haben Wissenschaftlerinnen und Wissenschaftler eine riesige genetische Komponentenliste der immunologischen Kommunikationswege erstellt. Wir kennen inzwischen unzählige Rezeptoren und Signalmoleküle der angeborenen Immunantwort. Zeugnis dessen sind Illustrationen, die diese Signal-Komponenten in verschlungenen Kaskaden darstellen und die ihrer Komplexität wegen bei Studierenden gefürchtet sind. Um Signalwege in ihrer Gesamtheit zu erfassen, mag diese Darstellungsform zellulärer Schaltpläne hilfreich sein – sie verdeckt jedoch Schwachstellen unseres Wissens: Die Schaltplan-Darstellung suggeriert eine räumliche Aufteilung und Abfolge, die wir oft gar nicht kennen. Interaktionen werden durch eine schlichte Abfolge von Pfeilen dargestellt, sollen aber eigentlich ein komplexes zeitliches und räumliches Geschehen abbilden. Im Gegensatz zu einem zweidimensionalen Schaltplan sind Zellen dreidimensional, in Kompartimente eingeteilt und – verglichen mit einem Signalmolekül – riesig.
Wo treffen sich Signalmoleküle in der Zelle?
Wenn wir Signalwege über ihre Komponenten hinaus verstehen wollen, müssen wir daher untersuchen, wo sich Signalmoleküle treffen, um eine Botschaft weiterzugeben und wie sie miteinander interagieren. Diesen Fragen gehen wir in meiner Arbeitsgruppe auf den Grund. Wir wollen grundlegende Mechanismen der Signalweitergabe verstehen und nutzen dazu hochauflösende Mikroskopie, um diese Mechanismen in Echtzeit zu beobachten.
Unsere Forschung konzentriert sich auf einen Mechanismus, dessen Rolle in der Zelle erst seit kurzer Zeit untersucht wird: Es handelt sich um die sogenannten Supramolekularen Organisationszentren, kurz SMOCs. An SMOCs bündeln sich Signalmoleküle wie in einer Schaltzentrale, sie können Signale der angeborenen Immunantwort empfangen und weitergeben. Möglicherweise helfen SMOCs auch dabei, den Schwellenwert für eine Immunreaktion zu regulieren, also zu bestimmen, ab welcher Signalstärke eine Immunreaktion ausgelöst wird.
Das Myddosome: Hier entsteht eine zelluläre Schaltzentrale
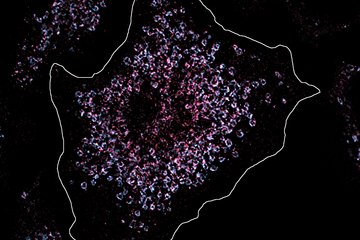
Um ihre Funktionsweise zu verstehen, haben wir ein spezifisches SMOC untersucht: das Myddosome, das sich an Rezeptoren der angeborenen Immunantwort in der Zellmembran bildet. Die Entstehung des Myddosome haben wir mit hochauflösender Mikroskopie verfolgt. Dazu haben wir Mikroskopie-Objektträger mit einer Lipid-Membran beschichtet, in die wir Interleukine eingebaut haben – Botenstoffe, an deren Rezeptoren sich das Myddosome bildet. So konnten wir eine Oberfläche herstellen, die bei Kontakt ein Signal in Immunzellen auslöst.
Um die Bildung des Myddosomes zu beobachten, haben wir die drei Komponenten des Myddosomes gefärbt: MyD88, IRAK4, IRAK1. Für die Färbung haben wir Immunzellen mit der Genschere CRISPR/Cas9 bearbeitet und die betreffenden Proteine mit fluoreszierenden Farbstoffen markiert. Unter dem Laserlicht eines Fluoreszenzmikroskops leuchten diese Farbstoffe.
Zellsignale unter dem Mikroskop: Live und in Farbe
So konnten wir live verfolgen, wie sich Myddosomes an der Zellmembran bilden. Mithilfe von Videos konnten wir den dynamischen Auf- und Abbau dieser Strukturen dokumentieren, sobald eine Zelle in Kontakt mit dem Botenstoff Interleukin kam. Dabei konnten wir zeigen, dass sich das Myddosome zunächst aus MyD88 Oligomeren formt – also vielen zusammenhängenden MyD88 Proteinen. Diese Strukturen sind zu Anfang oft instabil, wachsen und fallen wieder auseinander. Selbst mit ungeübtem Auge lässt sich dieser Prozess in unseren Videos beobachten.
Video: Akkumulation von MyD88-Oligomeren und nachfolgendes Signalling via IRAK
Je mehr sie wachsen, desto stabiler werden diese Oligomere, und große Oligomere, ab vier MyD88-Proteinen, rekrutieren dann IRAK4 und IRAK1 an die Zellmembran. Zusammen bilden diese Komponenten das Myddosome, das das extrazelluläre Signal der Interleukine innerhalb der Zelle weitergibt und eine Immunantwort anstößt. Die Größe des MyD88-Oligomers scheint also entscheidend für die Signalweitergabe zu sein: Erst wenn der Rezeptor ausreichend aktiviert wurde, die Gefahr durch eine Entzündung also wirklich gegeben ist, bündeln sich genügend MyD88-Proteine, um das Signal weiterzuleiten und eine Reaktion hervorzurufen. Dies wiederum könnte unnötige Reaktionen verhindern.
Grundmechanismen der zellulären Immunsignale
Wir haben ebenfalls einen möglichen Mechanismus gefunden, der eine überschießende Immunreaktion verhindern könnte: IRAK4 scheint nämlich auch eine regulierende Funktion zu besitzen. Wir haben Zellen erzeugt, in denen die Produktion von IRAK4 gentechnisch ausgeschaltet war. In diesen Zellen sind die MyD88-Oligomere immer weitergewachsen und haben schließlich Superoligomere gebildet. Übertragen auf die Signalweitergabe bedeutet dies eine unregulierte Aktivierung. IRAK4 aktiviert also nicht nur den nächsten Schritt in der Signalkette, sondern stoppt auch die weitere Oligomerisierung von MyD88.
Wir vermuten, dass der Selbst-Aufbau des Myddosomes ein wichtiger Schalter für Entzündungsreaktionen ist. Durch unsere Experimente können wir nun besser nachvollziehen, wie dieser Schalter durch die Menge an gebündelten MyD88-Proteinen gesteuert werden kann: Es kommt darauf an, wie viele Signalmoleküle wo vorhanden sind. Dieser Mechanismus ist nicht auf das Myddosome beschränkt und ist möglicherweise auf viele andere SMOCs übertragbar. Das könnte uns dabei helfen, die Grundprinzipien der Immunsignalwege zu verstehen – und dem zweidimensionalen Schaltplan der Zelle mehr Tiefe zu verleihen.