Forschungsbericht 2018 - Max-Planck-Institute für biophysikalische Chemie
Ein Schalter für menschliche Gene
Wie Gene aktiv werden
Damit sich ein Organismus entwickeln kann, müssen zu bestimmten Zeiten und an bestimmten Stellen des Embryos Teile der Erbinformation aktiviert werden. Die Erbinformation liegt in allen Zellen in Form von DNA vor und umfasst Zehntausende von Genen. Diese werden durch den Prozess der Gen-Expression aktiviert. Der erste Schritt dieses Prozesses, der zur Synthese von Proteinen führt, ist die Transkription. Während der Transkription eines Gens wird eine RNA-Kopie von einem DNA-Abschnitt erstellt. Die Transkription wird von Enzymen bewerkstelligt, den sogenannten RNA-Polymerasen. Um zu verstehen, wie Gene aktiviert oder abgeschaltet werden, ist es notwendig, die Struktur und Funktion der RNA-Polymerasen zu studieren. Diese Untersuchungen müssen sowohl im Reagenzglas (in vitro) wie auch in der lebenden Zelle (in vivo) durchgeführt werden.
Der Genkopierer heißt RNA-Polymerase II
In eukaryotischen Zellen gibt es mehrere RNA-Polymerasen, die verschiedene Arten von Genen kopieren. Unter diesen hat die RNA-Polymerase II eine herausragende Bedeutung, da sie alle Gene kopiert, die Bauanleitungen für Proteine enthalten. Proteine nehmen im Organismus die unterschiedlichsten Funktionen wahr; sie sind für alle Lebensprozesse von essenzieller Bedeutung [1]. Unsere Arbeitsgruppe konnte Bilder der molekularen Struktur der RNA-Polymerase II in vielen verschiedenen Zuständen erhalten und daraus Filme über den Transkriptionsvorgang erstellen. Nun können wir dem Kopiervorgang in atomarem Detail zusehen.
Wie der Beginn des Gens gefunden wird
Vor kurzem konnten wir sogar in drei Dimensionen darstellen, wie die Transkription beginnt [2]. Die RNA-Polymerase II benötigt dazu mehrere zusätzliche Proteinfaktoren; nur so kann sie den Startpunkt der Transkription finden, die DNA-Doppelhelix öffnen und die Synthese der RNA-Kopie beginnen. Die Strukturanalyse dieser Vorgänge ist äußerst schwierig, da viele Dutzend Proteinfaktoren beteiligt sind und der Prozess sehr dynamisch ist. Diese technischen Schwierigkeiten konnten durch eine Kombination verschiedener Methoden überwunden werden. Dabei spielen die Kryo-Elektronenmikroskopie, die Röntgenkristallografie und die Massenspektrometrie eine wichtige Rolle. Die dank dieser Techniken ermittelte 3D-Struktur der RNA-Polymerase II zeigt das Enzym eingebettet in einen sogenannten Initiationskomplex, und diese Strukturbilder ermöglichen viele Einblicke in den Mechanismus des Beginns der Transkription. So konnten wir etwa vorschlagen, wie einer der beteiligten Faktoren die DNA öffnet und ins aktive Zentrum des Enzyms lädt, wo sie als Matrize für die RNA-Synthese dient.
Wenn der Kopiervorgang stockt
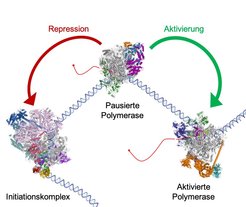
Nachdem die Transkription erfolgreich gestartet ist, bleibt die Polymerase allerdings oft gleich wieder stehen. Das Enzym pausiert dann ganz am Beginn des Gens und wartet auf weitere Signale der Zelle. Es ist schon lange bekannt, dass solche positiven Signale in der Lage sind, einen Austausch von negativen, hemmenden Faktoren gegen positive Faktoren zu bewerkstelligen und so dafür zu sorgen, dass die Polymerase bis ans Ende des Gens kopiert, sodass eine vollständige RNA-Kopie erstellt wird und nachfolgend das Gen in der Zelle aktiv ist. Wir konnten nun mithilfe der Strukturbiologie zeigen, wie die Polymerase pausiert und wie sie wieder aktiviert wird [3, 4]. Ein negativer Faktor (NELF) ist dazu in der Lage, den inaktiven Zustand der pausierten Polymerase zu stabilisieren: In diesem inaktiven Zustand sind die DNA und RNA im aktiven Zentrum des Enzyms nämlich nicht korrekt angeordnet. Mehrere positive, aktivierende Faktoren (P-TEFb, PAF, SPT6) können nun wiederum den negativen Faktor NELF ablösen und so eine hohe Aktivität der RNA-Polymerase II bewirken. Ein Vergleich der Molekülstrukturen der pausierten und aktivierten RNA-Polymerase II konnte dies sehr eindrucksvoll zeigen (Abb. 1).
Wie der Genschalter funktioniert
Trotz dieser detaillierten Einsichten blieb die Frage offen, wie es überhaupt möglich sein kann, dass Gene durch die Pausierung der Polymerase kontrolliert werden. Da die Pausierung oft nur einige Minuten umfasst, die RNA-Synthese aber oft Stunden dauert, ist es keineswegs klar, wie durch eine Änderung der Pausierung die Menge an RNA-Produkt maßgeblich geändert werden kann. Mit anderen Worten: Wie kommt es, dass kleine Änderungen in der Pausierung die Menge an synthetisierter RNA grundlegend modifizieren können? Wir konnten dieses Rätsel mithilfe verschiedener Methoden der sogenannten funktionalen Genomik in Zellen und mittels Bioinformatik aufklären [5]. Uns gelang der Nachweis, dass die pausierte Polymerase am Beginn des Gens die Initiation der Transkription limitieren kann. Genau aufgrund dieser Tatsache beeinflusst die Dauer der Pausierung der Polymerase die Anzahl derjenigen Initiationsereignisse, die in einem gegebenen Zeitraum an einem Gen stattfinden könnten. Das wiederum führt zu einer Änderung der Anzahl an RNA-Produkten und somit der Genaktivität. Dieser Mechanismus der Genkontrolle ist es, der wie ein Schalter für Gene wirkt; er geschieht besonders oft während der Differenzierung von Zellen in verschiedene Zelltypen. Zukünftig können wir nun, basierend auf dieser Erkenntnis, die Aktivierung von Genen während der Zelldifferenzierung studieren.
