Forschungsbericht 2017 - Max-Planck-Institute für biophysikalische Chemie
Molekulare Auflösung in der optischen Mikroskopie
Vom Mikro-Skop zum Nano-Skop
Es ist der „Heilige Gral“ der Lichtmikroskopie: die Trennschärfe dieser Methode so weit zu verbessern, dass man dicht benachbarte Moleküle einzeln auflösen kann. Mit einem neuen fluoreszenzmikroskopischen Verfahren, MINFLUX genannt, lassen sich erstmals Moleküle trennen, die nur Nanometer (millionstel Millimeter) voneinander entfernt sind. Dies ist die ultimative Grenze dessen, was in der Fluoreszenzmikroskopie möglich ist. Das innovative „Nanoskop“ ist mehr als einhundert Mal schärfer als herkömmliche Lichtmikroskope und übertrifft selbst die bisher besten fluoreszenznanoskopischen Methoden – das zuerst entwickelte STED (Stimulated Emission Depletion) und die später erfundenen PALM/STORM-Verfahren (Photo-Activated Localization Microscopy/Stochastic Optical Reconstruction Microscopy) – um das bis zu 20-fache. MINFLUX nutzt die jeweiligen Stärken von STED und PALM/STORM in einem völlig neuen Konzept und hat das Potenzial, ein fundamentales Werkzeug der Zellbiologie zu werden. Dieses Verfahren wird es nämlich in Zukunft erlauben, Zellen molekular zu kartografieren und schnelle Vorgänge in ihrem Inneren in Echtzeit sichtbar zu machen, was unser Wissen über die molekularen Abläufe in lebenden Zellen revolutionieren könnte.
Der Nobelpreis für Chemie 2014 würdigte das Vordringen der fluoreszenzmikroskopischen Auflösung in die Dimension einzelner Moleküle – unter klassischer Verwendung von fokussiertem Licht und normalen Objektiven. Zwar hatte der Physiker Ernst Abbe 1873 formuliert, dass die Auflösung von Lichtmikroskopen auf die halbe Wellenlänge des Lichts begrenzt ist – das sind etwa 200 Nanometer. Und auch mehr als einhundert Jahre später behält dieses Abbe-Limit physikalisch seine Gültigkeit. Doch die 1994 vorgeschlagene und fünf Jahre später experimentell umgesetzte STED-Mikroskopie hatte gezeigt, dass sich diese Grenze überwinden lässt. STED und das ein paar Jahre danach entwickelte PALM/STORM erreichen in der Praxis eine Trennschärfe von typischerweise etwa 20 bis 30 Nanometern – also rund zehn Mal besser als das Abbe-Limit.
Die Stärken von STED und PALM/STORM kombinieren
Sowohl STED als auch PALM/STORM trennen benachbarte fluoreszierende Moleküle, indem sie diese nacheinander an- und ausschalten und so sequenziell zum Leuchten bringen. Die beiden Methoden unterscheiden sich aber in einem wesentlichen Punkt: Die STED-Mikroskopie setzt einen Donut-förmigen Laserstrahl ein, um das Leuchten der Moleküle an genau festgelegten Koordinaten in der Probe zu unterdrücken. Der Vorteil: Durch den definierten Donut-Strahl weiß man präzise, an welchem Punkt im Raum sich das gerade leuchtende Molekül befindet. Der Nachteil: Den Laserstrahl kann man in der Praxis nicht derart intensivieren, um nur ein einziges Molekül anzusteuern. Bei PALM/STORM hingegen erfolgt das An- und Ausschalten an zufälligen Orten Molekül für Molekül mit dem Vorteil, dass man bereits auf der Ebene einzelner Moleküle arbeitet, aber auch dem Nachteil, dass man deren genaue Positionen nicht kennt und erst herausfinden muss. Im Experiment lässt sich auf diese Weise routinemäßig noch keine molekulare Auflösung erreichen.
Der Negativ-Test, oder: Auch die Abwesenheit von Signalen ist verwertbare Information
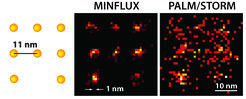
Abb. 1: Mit dem MINFLUX-„Nanoskop“ kann man erstmals Moleküle optisch trennen, die nur wenige Nanometer voneinander entfernt sind. Links ist schematisch die Anordnung fluoreszierender Moleküle (gelbe Kugeln) dargestellt. Während PALM/STORM bei gleicher Molekül-Helligkeit nur ein diffuses Bild liefern kann (hier in einer Simulation unter idealen technischen Bedingungen), ist die Anordnung der Moleküle mit dem MINFLUX-Nanoskop (Mitte) klar zu erkennen.
Die Stärken beider Techniken können in einem neuen Konzept vereint werden: MINFLUX (Minimal Photon Fluxes, minimale Flüsse von emittierten Photonen) schaltet, wie PALM/STORM, einzelne Moleküle zufällig an und aus. Gleichzeitig bestimmt es aber, wie STED, deren exakte Position mit einem Donut-förmigen Laserstrahl, der im Gegensatz zu STED aber nicht zum Abregen, sondern zum Anregen der Fluoreszenz benutzt wird [1]. Liegt das Molekül auf dem Donut-Ring, so leuchtet es; liegt es exakt in seinem dunklen Zentrum, so leuchtet es zwar nicht, man hat jedoch seine genaue Position gefunden (Abb. 1). Damit diese Position mit höchster Präzision und schnell ermittelt werden kann, nutzt ein ausgeklügelter Algorithmus zur schnellen Positionierung des Donut-Laserstrahls das volle Potenzial der Information, die in den kleinen detektierten Molekülsignalen nahe des Zentrums, oder mit anderen Worten: des Intensitäts-Minimums steckt.
Einhundertmal bessere Zeitauflösung
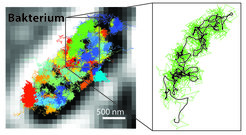
Neben der molekularen Auflösung bietet die Kombination von STED und PALM/STORM einen weiteren großen Vorteil. MINFLUX ist im Vergleich sehr viel schneller. Da die Technik mit dem Donut-Laserstrahl arbeitet, kommt sie mit wesentlich weniger Lichtsignal, das heißt Fluoreszenz-Photonen, pro Molekül als PALM/STORM aus. Bereits mit STED konnte man Echtzeit-Videos aus dem Inneren lebender Zellen aufnehmen. Doch nun lassen sich die Bewegungen von Molekülen in einer Zelle mit einer einhundert Mal besseren zeitlichen Auflösung verfolgen. Dank MINFLUX war es beispielsweise in ersten Experimenten möglich, die Bewegung von Molekülen in einem lebenden Escherichia coli-Bakterium in bisher unerreichter Zeitauflösung zu „filmen“ (Abb. 2).
Was die Geschwindigkeit molekularer Bewegungsmuster angeht, sind die Möglichkeiten von MINFLUX noch längst nicht ausgereizt. Zukünftig werden sich selbst extrem schnelle Abläufe in lebenden Zellen untersuchen lassen – etwa die Bewegung zellulärer Nanomaschinen oder gar die Faltung von Proteinen.
Literaturhinweise
Science 355, 606-612 (2017)

